Technical Data
技术资料
本文基于2018年截至11月的Molecular & Cellular Proteomics 对DIA数据提交的意见稿,并针对Spectronaut用户进行专门说明。第一部分为手稿提交时对文章材料方法部分撰写的要求及参考示例,第二部分为数据上传公共存储平台的操作流程(以ProteomeExchange的联盟网站,中国国家蛋白中心建立的iProX数据库为例)。

1. 材料方法撰写要求
1.1 实验部分
1.1.1 实验设计和统计描述:
作者必须在实验方法部分添加一个subsection标题命名为 “Experimental Design and Statistical Rationale”. 该部分必须明确注明以下信息:
1)样品的数量和类型;
2)用于分析和结果中展示的样本数量;
3)该实验所包含的技术重复生物学重复以及过程重复的数目,如果没有设置任何重复,需明确给出该结果可被接受的理由;
4)样本数目及重复数目设置的合理性;
5)是否采用了标准肽段或蛋白矫正保留时间;
6)样本采集顺序随机化的方法;
7)是否建立真实谱图库,建库采用的样本的数量和类型(包括是否设置生物学重复/技术重复);
8)描述数据处理和统计学分析所采用的算法或者程序。需完整描述采用或者引用用于后续数据统计分析的方法并阐述采用该方法的合理性。
示例:
Experimental Design and Statistical Rationale
Two group tissue samples (e.g. liver, brain) were selected, and each group is represented by nine biological replicates. Nine individuals per group also permits sufficient sample size for standard statistical tests such as two-way ANOVA and PCA and so on, because the variation between technical replicates in DIA is less than the difference between biological replicates (technical error is ~1/3 of biological error [1]). For library generation by DDA, all 18 samples were pooled as a mixture and fractionated by high pH separation with 10 fractions. And all 18 samples were processed by DIA individually to assess theproteome differences. MS1 and MS2 data were all acquired, and samples acquisition by random order. The iRT kit (Ki3002, Biognosys AG, Switzerland) was added to all of the samples to calibrate the retention time of extracted peptide peaks. The statistical analysis of the DIA dataset was performed by Spectronaut X (Biognosys AG, Switzerland) including data normalization and relative protein quantification. After Student’s t-Test, different expressed proteins were filtered if their Qvalue<0.05 and="" absolute="" avg="" log2="" ratio="">0.58.
Reference:
[1] Williams, E.G., et al., Systems proteomics of liver mitochondria function. Science, 2016.
352(6291): p. aad0189.
1.2 数据采集
DIA数据采集的方法有很多种,作者需要给出其认为有利于评估此检测结果的所有参数,包括:
1)是否采集了MS1数据及扫描范围;
2)是否进行了分级,如果分级那么分级相关的参数,窗口的数目,是否设置了重叠窗口以及循环时间等。
示例:
DIA data acquisition
Data-independent Acquistion (DIA): The peptides were re-dissolved in solvent A (A: 0.1% formic acid in water) and analyzed by on-line nanospray LC-MS/MS on an Orbitrap Q Exactive HF coupled to EASY-nLC 1200 system (Thermo Fisher Scientific, MA, USA). 3 μL peptide sample was loaded to analytical column (Acclaim PepMap C18, 75 μm x 25 cm) and separated with 120 min gradient, from 5% to 35% B (B: 0.1% formic acid in ACN). The column flow rate was maintained at 200 nL/min. The electrospray voltage of 2 kV versus the inlet of the mass spectrometer was used. The mass spectrometer was run under data independent acquisition mode, and automatically switched between MS and MS/MS mode. The full scan was performed between 350–1,600 m/z at 60,000 resolution. The automatic gain control target for the MS scan was set to 3e6 and the maximum injection time was 20 ms. The MS/MS scan was performed at 30,000 resolution (automatic gain control target of 1e6 and auto for injection time. The collision energy was 27, and stepped collision energy was 5%. DIA was performed with variable Isolation window, and each window overlapped 1 m/z, and the window number is 42, total cycle time is 3s.
DIA Raw Data analysis
Raw Data of DIA were processed and analyzed by Spectronaut X (Biognosys AG, Switzerland) with default settings, Retention time prediction type was set to dynamic iRT. Decoy generation was set to mutated which similar to scrambled but will only apply a random number of AA position swamps (min=2, max=length/2). Interference correction on MS2 level was enabled. The false discovery rate (FDR) was set to 1% at peptide level. After Student’s t-Test, different expressed proteins were filtered if their Qvalue<0.05 and="" absolute="" avg="" log2="" ratio="">0.58. The DIA raw data and the results reported at protein level as well as peptide level is available at iPROX(or supplement)
1.3 DIA数据的分析方法
DIA数据的分析大致可以分为两种策略;一种是试图将肽与单个谱图(以谱图为中心,Spectrum-Centric)匹配,另一种是试图检测数据文件中某个给定的肽(以肽为中心,Peptide-Centric)。对于两种策略,都可以匹配蛋白质数据库或谱图库,但是大多数以谱图为中心的分析查询蛋白质数据库而以肽为中心的方法主要使用谱图库。下面就根据不同的分析策略分别进行指导,有些工作可能使用这两种方法的组合,在这种情况下,这两套指导方针都是适用的。
1.3.1 Spectrum-Centric策略进行的DIA分析
(1)峰列表(peak list)生成:
a.说明用于创建峰列表的方法和/或程序(包括版本号和/或日期)。
b.列举创建此峰列表时使用的参数,特别是可能影响后续数据库搜索质量的任何处理。例如平滑、信噪比阈值,带电荷状态或去同位素峰,去多重累积,肽的不同电荷状态(m/z或者漂移分离)对产物离子谱的相对贡献。
c.描述在峰列表文件中是如何分配离子的保留/漂移时间和强度的。
d.说明一个检测到的碎片离子可以包含的母离子峰的最大数量。
e.如果在创建峰列表时执行了额外的自定义处理,例如聚类或过滤,则应引用所采用的方法和/或程序(包括版本号)。
f.搜索引擎:必须提供用于数据库搜索的所有程序的名称和版本(或发布日期)。
g.序列数据库或谱图库:必须列出所使用的所有序列数据库或谱图库的名称和版本(或发布日期)。如果是自建的序列数据库或者谱图库,则必须完整的描述序列或者谱图的来源以及用于建库的软件。必须说明从每个序列数据库或者谱图库中搜索到的实际条目数。如果使用的数据库或谱图库非常小(< 1000个条目)或排除了常见的污染物则必须提供明确的理由,因为这可能会产生错误的匹配和不准确的错误发现率(FDR)。
h.酶特异性:必须完整描述酶切过程所采用的所有蛋白酶种类,包括漏切的数量和非特异性酶切(如semitryptic),必须列出。
i.固定修饰:所有修饰的列表(包括特异性残基)。
j.可变修饰: 所有修饰的列表(包括特异性残基)。如果没有指定固定或可变的修饰,也须说明。
k.母离子及碎片离子的质量容差(如果是自定义设置的需要说明这一点;一些软件会自动确定这一点)。
l.已知污染物排除:所有鉴定出的污染物的谱峰是否被排除(或是否这些碎片离子被应用到校准过程)。
m.阈值评分/期望值:被用于谱图鉴定的标准以及理由需明确陈述。
n.肽段、蛋白质鉴定的错误发现率(FDR)和批处理水平:对于大规模实验,任何能用来评价数据鉴定准确性的额外的统计分析结果,或错误发现率的计算,如反库检索结果或其他计算方法的结果需进行描述。
示例:
Peak List Generation
Spectronaut X (Biognosys AG, Switzerland) with default settings was used to generate a peak list by the database of swissprot homo sapiens 201803. Trypsin was assumed as the digestion enzyme. Missed Cleavages was set to 2, and the decoy database was generated by reversed sequence. Carbamidomethyl (C) was specified as the fixed modification. Oxidation (M) and Acetyl(Protein N-term) was specified as the variable modifications. Kernel Density Estimator was performed to calculate the Pvalue, because it usually provides the best fit for estimating null-distribution Pvalues. Qvalue (FDR) cutoff on precursor and protein level was applied 1%. Remove the peptide if there are not at least 3 fragment ions, and kept best 6 fragments per peptide. All selected fragment ions passing the filters are used for quantification. The average top 3 filtered peptides which passed the 1% Qvalue cutoff were used to calculate the major group quantities. 34,767 precursors, 26,538 peptides, 3,758 proteins and 3,650 protein groups were searched. After Student’s t-Test, different expressed proteins were filtered if their Qvalue<0.05 and="" absolute="" avg="" log2="" ratio="">0.58. The database and reports are available on iPROX (or supplement) (项目号)
1.3.2 Peptide-Centric 策略的DIA分析
谱图库:
对于所有谱图库,必须报告谱图的数量和它们所覆盖的蛋白质的数量(target和Decoy)。对于较小的库(小于1000个条目),必须提供合理的理由。
(1)谱图库的建立是否作为本研究的一部分:
a.如果从DDA数据创建谱图库,则DDA MS/MS的创建过程必须完整。
b.用于建库的软件 (包括版本号)
c.多个谱图可以被一个肽段使用的标准;
d.如果一个谱图被添加到谱图库中,选择的标准是什么,例如最佳评分,最可信的修饰位点。
e.如果谱图库中创建了合成谱图,那么合并谱图所采用的参数也需要说明
f.是否只有一个肽段用于创建库,即是否去除未修饰或修饰的肽段
g.某些峰(如前体离子)是否从谱图库中删除
h.谱峰是否卡了阈值(例如最低信噪比,每张谱图最大的谱峰数目)
i.计算的得出的谱图库中每个条目的FDR值;包括计算方法。分析结果是否整合了多种算法的分析结果,如果是,那么是使用的什么软件/方法来进行FDR的控制的。
(2)如果使用的是公开的谱图库:
a.库的版本号。提供文献引文。
b.数据库可以获得/下载的位置。
c.被额外利用的数据库中的图谱元数据有哪些;例如保留时间,离子迁移率。
d.是否对库进行进一步处理;例如进一步参数调整;峰阈值修改。
(3)如果使用的是预测的谱图库:
a.用于建库的软件(包括版本号)
b.建库的参数包括(如蛋白质序列的来源;酶特异性假定;包括哪些修饰;肽段长度/质量范围等)。
c.如果谱图库存在Decoy库:
d.有多少Decoy条目(相对于target条目的比例)
e.如何分配这些Decoy蛋白(允许蛋白质水平的FDR估计)?
f.Decoy蛋白的谱图是如何产生的?
g.利用谱图库进行数据库检索:
h.用于peptide-centric分析的软件的名称和版本号
i.是否采集母离子?
○如果是,母离子信息是如何使用的?
○母离子匹配的质量容差是多少?
j.保留时间或离子迁移率是否用来辅助定性?
○如果是,如何进行的;例如是否利用了预测时间/移动的窗口?
○利用什么方法来进行保留时间的对齐,或者描述用来评估保留时间重现性的方法。
k.色谱峰的形状是否作为一个参数用于结果评分?如果是,怎么做的?
l.有多少质谱峰被用于识别一个肽段?(对于某些软件,这可能是一个范围)
○这些谱峰挑选的标准是什么;例如,在谱图库中的相对强度,必须大于某一分子量;必须在一定的质量范围内等
m.搜库时碎片离子的质量容差。
n.如果是修饰位点的鉴定,用于判断修饰位点可靠性的方法
o.肽段、蛋白质鉴定的错误发现率(FDR)和批处理水平:对于大规模实验,任何能用来评价数据鉴定准确性的额外的统计分析结果,或错误发现率的计算,如反库检索结果或其他计算方法的结果需进行描述。
示例:
Library Generation
Data-dependent Acquistion (DDA): The peptide mixture was re-dissovled in the buffer A (buffer A: 20 mM ammonium formate in water, pH 10.0, adjusted with ammonium hydroxide), and then fractionated by high pH separation using Ultimate 3000 system (ThermoFisher scientific, MA, USA) connected to a reverse phase column (XBridge C18 column, 4.6 mm x 250 mm, 5 μm, (Waters Corporation, MA, USA)). High pH separation was performed using a linear gradient, starting from 5% B to 45% B in 40 min (B: 20mM ammonium formate in 80% ACN, pH 10.0, adjusted with ammonium hydroxide). The column was re-equilibrated at the initial condition for 15 min. The column flow rate was maintained at 1 mL/min and the column temperature was maintained at 30℃. Ten fractions were collected; each fraction was dried in a vacuum concentrator. And then peptides were re-dissolved in 0.5% formic acid in 5% ACN) and analyzed by on-line nanospray LC-MS/MS on an Orbitrap Q Exactive HF coupled to EASY-nLC 1200 system (Thermo Fisher Scientific, MA, USA). 3 μL peptide sample was loaded to analytical column (Acclaim PepMap C18, 75 μm x 25 cm) and separated with 120 min gradient, from 5% to 35% B (B: 0.1% formic acid in 80% ACN). The column flow rate was maintained at 200 nL/min. The electrospray voltage of 2 kV versus the inlet of the mass spectrometer was used. The mass spectrometer was run under data dependent acquisition mode, and automatically switched between MS and MS/MS mode. The full scan was performed between 350–1,600 m/z at 60,000 resolution. The automatic gain control target for the MS scan was set to 3e6 and the maximum injection time was 50 ms. The dynamic exclusion was set to 30 s. The MS/MS scan was performed at 15,000 resolution (automatic gain control target of 5e5 and 60 ms maximum injection time. The collision energy was 30.
Spectral Library generation: Raw Data of DDA were processed and analyzed by Spectronaut X (Biognosys AG, Switzerland) with default settings to generate an initial target list, which contained 34,767 precursors, 26,538 peptides, 3,758 proteins and 3,650 protein group. Spectronaut was set up to search the database of swissprot homo sapiens 201803 assuming trypsin as the digestion enzyme. Carbamidomethyl (C) was specified as the fixed modification. Oxidation (M) was specified as the variable modifications. Qvalue (FDR) cutoff on precursor and protein level was applied 1%. The complete assay library including all relevant metadata is available at iPROX (or supplement)
1.4 结果部分
1.4.1 肽和蛋白质报告(见Spectronaut 导出的Report列表)
根据研究的重点,结果可能在肽段或蛋白水平上得到最适当的展示。必须提供一份结果列表,可以放在原稿中,或者如果结果文件很大,也可以作为补充材料随原稿一起提交给杂志。
对于蛋白质水平报告的结果,此表必须包括:
a.蛋白质的accession号
b.每个蛋白对应的肽段数目:计算这个数字时,氨基酸序列相同的多个匹配肽应该算作不同的肽,包括一个肽段不同的带电状态或者修饰状态。任何替代假设都必须是合理的。
c.如果某个蛋白被鉴定到了,那该蛋白在谱图库中的肽段数目是多少。
d.对于某些只鉴定到一个肽段的蛋白,肽段水平的信息必须提供,以及质谱和色谱信息 (哪个更合适;见下文)
e.对于肽段水平报告的结果,结果表必须包括:
f.蛋白质的accession号
g.所有匹配到的肽段序列。
h.母离子电荷数,以及检测到的m/z(质荷比,如果使用MS1数据)。
i.所有检测到的修饰。
j.对于peptide-centric分析,匹配和没有匹配到的碎片离子的数量和评估鉴定质量的统计结果。
k.对于spectrum-centric分析,打分,和/或肽段匹配的统计度量。
l.如果鉴定到的肽段包含修饰,衡量修饰可靠性的方法必须报告(或必须表明修饰的可靠性没有评估)。
m.如果报告鉴定到的肽段带有翻译后修饰,或者鉴定到的蛋白是基于一个唯一肽段(此定性结果不推荐)那么这些肽段对应的质谱图和色谱图都必须是可获得的。可以通过以下方式提供:
○把所有的数据和搜库结果上传到一个配备有查看功能的公共数据存储平台,这种方法优于直接发送给杂志。
或者
○提交的数据和搜索结果的文件格式允许一些免费的软件对谱图进行可视化查看。
请参见
http://www.mcponline.org/site/misc/annotated_spectra.xhtml
了解如何通过不同的软件实现谱图的注释。
Spectronaut分析结果可以导出为.sne文件。将该文件上传至公共数据存储平台即可。
一般在结果部分,我们鼓励作者展示在质谱数据中鉴定到的总离子的比例,该百分比可以通过使用任何软件对数据进行解释并且应说明如何确定或估计出该比例的。
1.4.2 定量
提供基于质谱分析的定量蛋白质组学结果的手稿必须提供以下信息:
1)所有相关的定量数据(作为肽段和蛋白鉴定列表的一部分),以及描述是如何通过原始数据得到这些定量值的(例如利用MS1还是MS2定量)。
2)后处理步骤的完整描述,如离群值剔除,通过鉴定打分值或者CV值进行数据过滤,通过阈值排除数据(例如,基于信噪比或最低离子数目。)
3)每个蛋白质用于定量的肽段数目(如果与定性采用的数目不一致)。
4)描述如何通过技术重复以及统计学方法来验证测量的分析方法的可靠性的。可能会引用一些标准方法或者特殊软件。然而,有必要证明手稿中包含的数据确实符合模型的假设。
5)描述如何通过生物学重复,统计方法来验证生物学可靠性的。单个样本的单次实验通常是不能接受的(除了作为测试生物信息学系统的数据)。如果生物学重复的来源相同是不能接受的 (例如,疾病样本),为了得到一个可靠的结论必须进行足够数量的类似生物样本的检测,并进行适当的证明。
6)描述定量过程中对共流出肽段的干扰是如何处理的
7)如果鉴定到修饰,那么是采用何种软件评估修饰位点鉴定的可靠性的。
8)正确估计不确定性和误差分析的方法。
9)对大量蛋白和多肽进行定量通常需要使用某种形式的多重假设检验校正。尽可能的应该为每个单独的蛋白质定量的可信度进行评估而不是基于全局数据集的评估。从手稿中的定量数据中得出的任何结论或假设,都必须与评估的不确定度估计值一致。
10)描述由多个亚型蛋白构成的蛋白组(protein group)的定量方式。
示例:
Quantification
Data extraction was determined by Spectronaut X based on the extensive mass calibration. Spectronaut Pulsar X will determine the ideal extraction window dynamically depending on iRT calibration and gradient stability. Qvalue (FDR) cutoff on precursor and protein level was applied 1%. All selected fragment ions passing the filters are used for quantification.MS2 interference will remove all interfering fragment ions except for the 3 least interfering ones. The average top 3 filtered peptides which passed the 1% Qvalue cutoff were used to calculate the major group quantities. After Student’s t-Test, different expressed proteins were filtered if their Qvalue<0.05 and="" absolute="" avg="" log2="" ratio="">0.58. The quantification report is available on iPROX (or supplement)
1.5 数据提交到公共存储库
所有由质谱输出的原始文件必须在首次提交手稿时存放在一个可公开访问并且不受作者控制的第三方数据存储平台(例如ProteomeXchange旗下的数据库iPROX)中。如果谱图库是作为研究的一部分创建的,那么用于创建谱图库的原始数据也必须上传(除非它已经公开可用,在这种情况下应该提供可供下载的位置),以及创建的谱图库(target库和Decoy库)。谱图库数据应优先作为单独的提交文件存放,以便更容易引用。存储库通常需要用户名和密码才能访问提交的数据集。这些信息必须在提交稿件给杂志时一同提供给编辑,并作为评审过程的一部分提供给审稿人。如果读取原始数据的软件不是被广泛应用的软件的话,我们鼓励将数据转换为mzML等开放格式(Thermo Fisher质谱仪器产生的数据无需这么做)。我们期望以尽可能接近原始数据的形式上传谱图数据,以免某些处理影响后续的数据解释度。
此外,必须提交一个文件作为补充材料(并且提交到原始数据的存储库),该文件映射每个原始数据文件、中间处理文件和结果文件之间的关系,并确定哪些是生物、技术或过程重复。所有的软件分析都必须记录所用软件的相应版本。
如果需要延迟或者不提交数据,必须在投稿时以书面形式提交请求给数据管理编辑[chalkley@cgl.ucsf.edu]。并且数据必须在出版时向公众提供。
有关此要求的进一步信息,请联系
mcp@asbmb.org。
2. 提交至公共数据库的操作流程
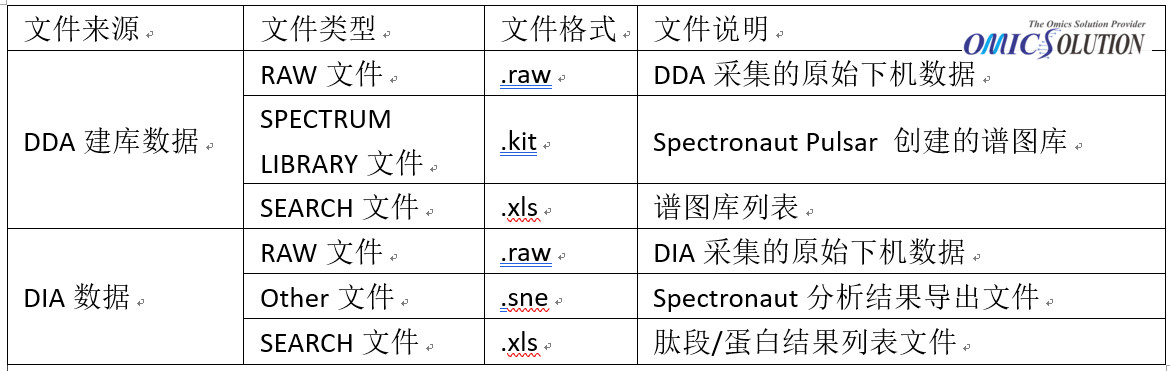
首先准备好需要上传的各种文件,所需文件如下:
iProX(http://www.iprox.org/),是一家在中国建立的蛋白质组学数据与知识中心,旨在促进蛋白质组学资源在世界范围内的共享。iProX目前由一个蛋白质组数据提交系统和一个蛋白质组数据库组成,其中前者遵照国际蛋白组学共享联盟(ProteomeXchange)的数据共享政策而建立。注册用户可以以公开或私有两种方式向iProX提交数据,一旦相关论文发表后,数据集将自动公开。
第一步,输入网址链接进入主页,点击网页右上角的“登录”输入账号和密码进入,如果是第一次登录需要点击“注册”,完善个人信息并提交申请获取账号和密码:

第二步,登录账号,创建项目,两种创建途径可以选择:①“提交”→“创建项目”或②“项目”→“New Project”

第三步,完善项目信息,其中“项目ID”及“PXID”是系统自动生成的,不需要填写,带“*”的为必填项:
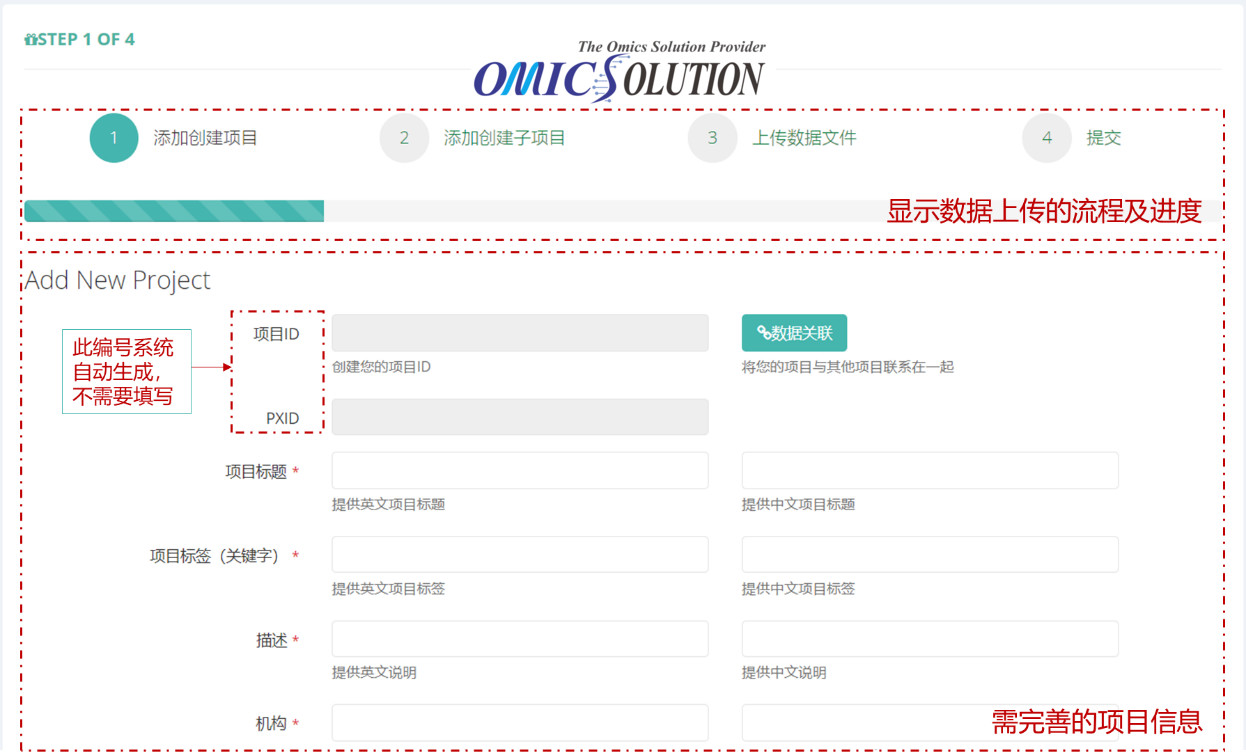
其中,“项目标签”即关键词;“描述”需要简要填写项目的描述,类似于文章中的摘要。
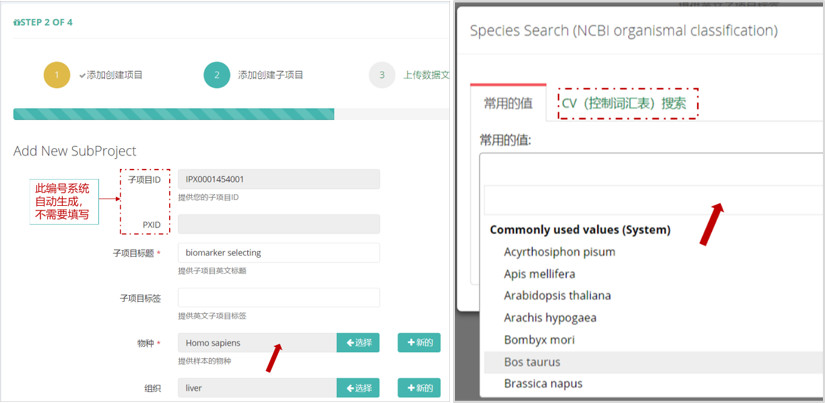
第四步,创建子项目,并按照提示填写数据集详细信息,点击对应的灰色选框,可以选择相应的物种,定量方法,酶解方法等信息。点击“常用的值”可以查找有无对应的参数,如果有可以直接选择,如果没有,则点击“CV(控制词汇表)搜索”输入关键词进行查找并选择:

其中,定量方法选择”SWATH MS”;实验类型选择“SWATH MS (Data-independent acquisition)”;酶解方法、PTMs、MS工具参照报告中设置的参数;其他信息及总结虽然不是作为必填项,但是建议尽可能的填写完整,以确保数据审核能顺利通过。“其他信息”可以简要描述实验步骤,包括酶解、分级、富集(针对翻译后修饰项目)、质谱、数据分析等过程,可以参考报告中的实验部分的描述。“总结”可以简要概括检测结果,包括鉴定到的谱图数,肽段数,蛋白数以及定量到的蛋白等信息。
第五步,完善中文信息,将上述信息翻译成中文即可:
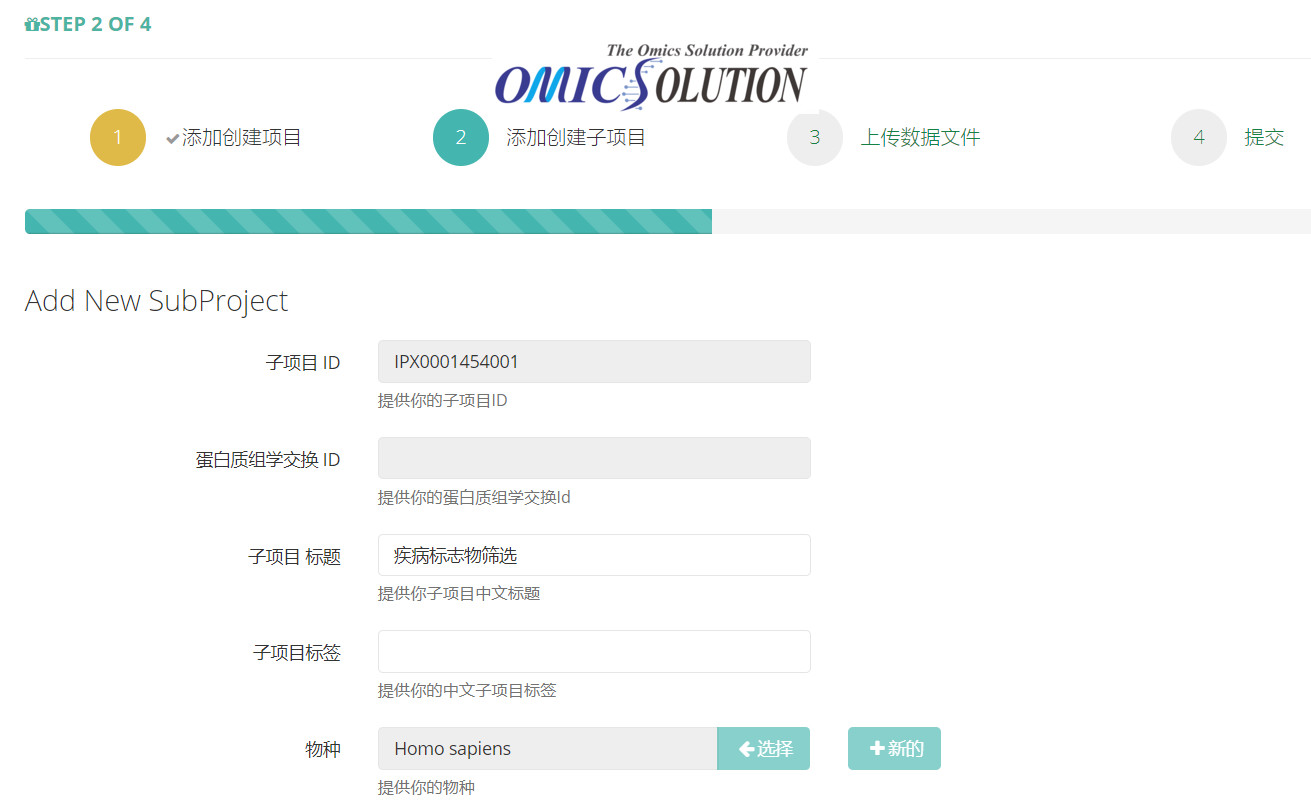
第六步,上传数据文件,将准备好的数据文件按照开始表1中对应的文件类型上传至平台,注意文件类型(File type)是否准确。选择“部分提交(Partial Submission)”(注:如果是中文网页,选择[PCV010.partial2])并且“通过Aspera上传”(文件小于4G可以选择通过浏览器上传)
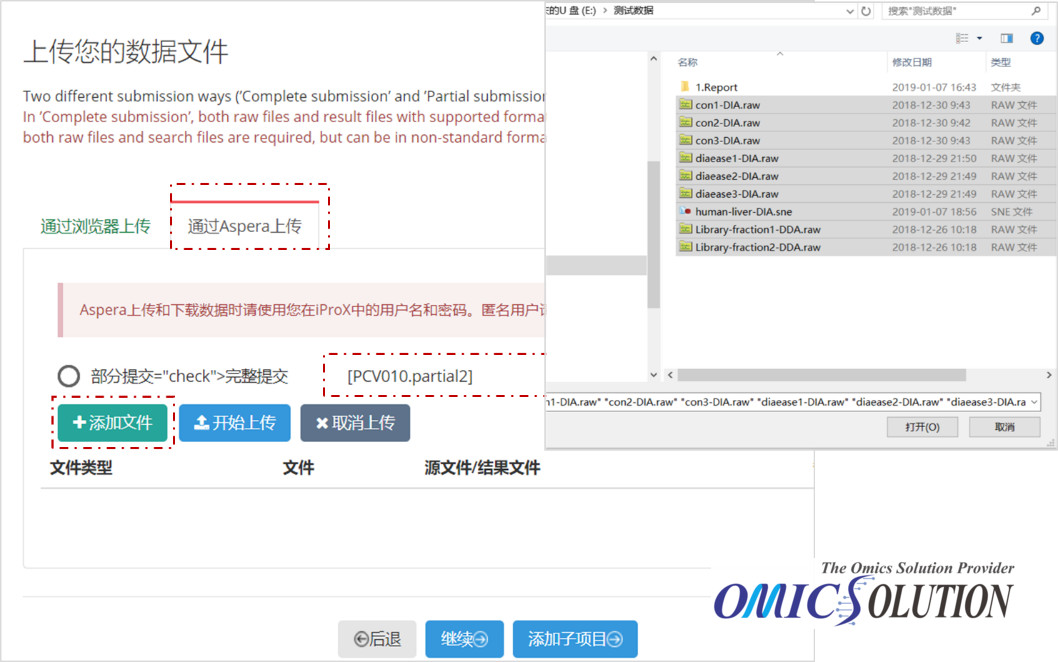
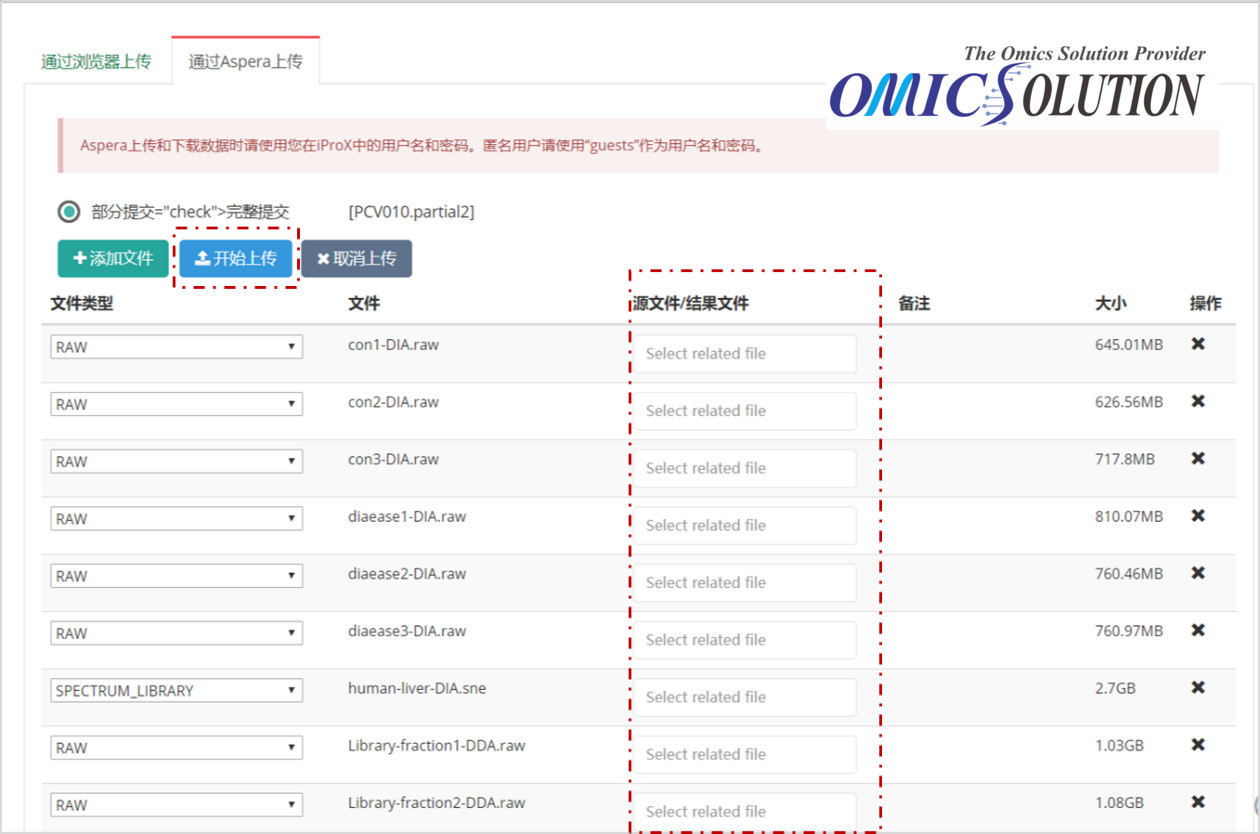
第七步,点击“Relative”为每一个文件添加一个或多个关联文件,完成上述操作后请点击“Start upload”进行文件上传,文件上传完成后页面上会出现提示,然后可以点击“Continue”进入提交页面,或者点击“Add New Subproject”再创建一个子项目:
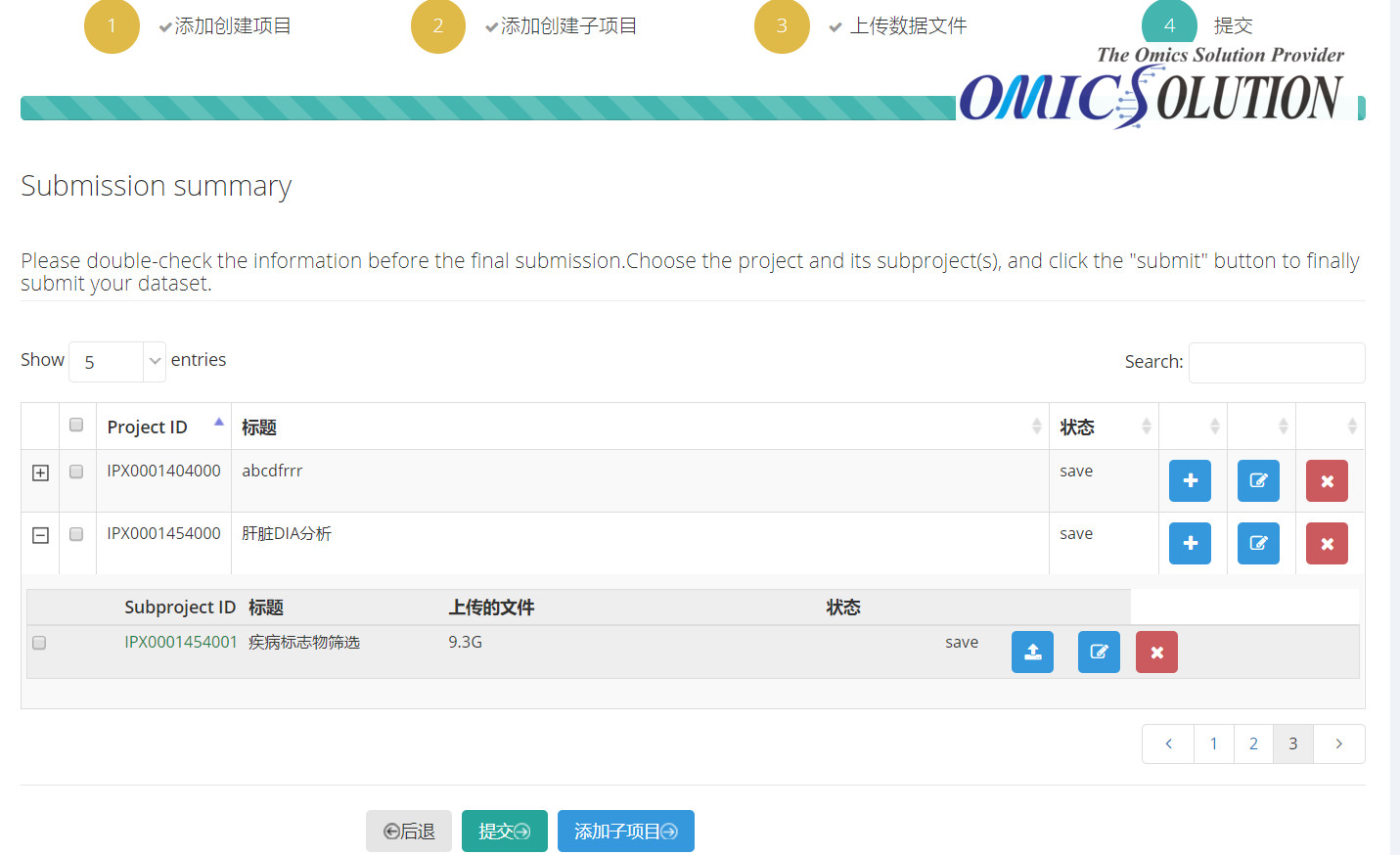
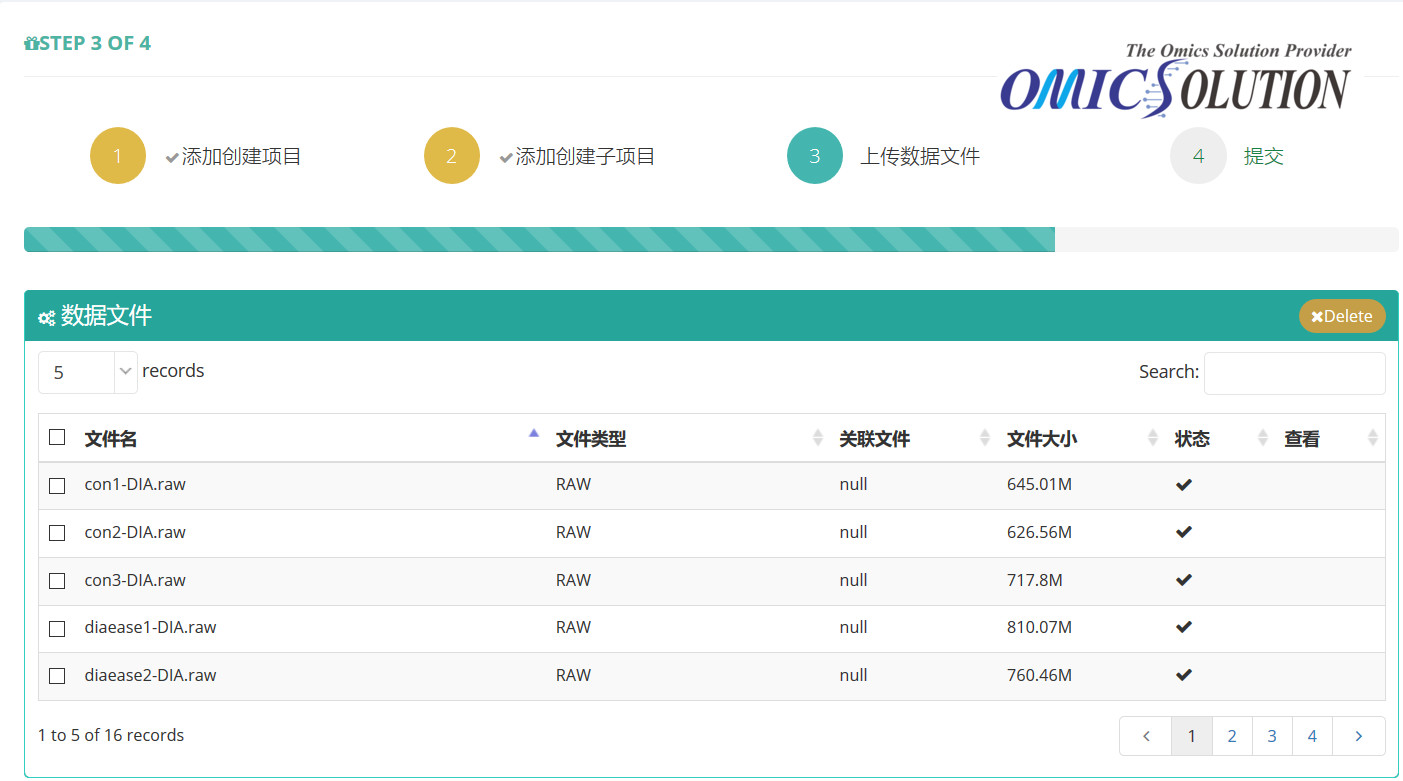
第八步,数据文件上传完成后,在网页上方可以浏览已上传的文件,如果上传错误可以选中文件,点击“Delete”删除,确认无误点击“继续”进入到提交页面。或者通过“我的项目“→“项目树”,选定项目/子项目名可在右侧查看填写好的项目/子项目信息,点击右侧上方的功能键进行相应操作:New Subproject在项目下添加新子项目,Edit进行项目/子项目信息修改页面,Upload在项目下上传新文件,delete删除save状态的项目/子项目:
第九步,确认项目/子项目信息无误后,点击页面下方submit进入数据提交页面,选择要提交的项目(该项目下所有子项目会自动选择,子项目和项目必须一起提交,享有相同的发布时效和被访问权限),点击下方“submit”键后项目(包括子项目)将提交给iProX数据管理员进行发布前审核,审核通过后提交者邮箱和站内信将收到相应通知,此时用户上传的数据文件正式完成提交。发送邮件反馈审核结果。