
Technical Data
技术资料
Data-independent acquisition-based SWATH-MS for quantitative proteomics: a tutorial
原作者:Christina Ludwig ,Ludovic Gillet,George Rosenberger ,Sabine Amon,Ben C Collins & Ruedi Aebersold
翻译:易算生物,沈诚频
摘要:许多个性化医疗、药物筛选或系统生物学领域的研究课题高度依赖于能对海量生物学个体样品进行准确而高度一致的定量蛋白质组学数据。SWATH-MS是DIA(数据非依赖采集技术)的一个特定子类,目前已逐步展现出能够同时满足蛋白质组深度覆盖和定量高准确性、高一致性要求的能力。在一次SWATH-MS检测中,样品中质量在一定范围内的所有多肽被系统性的、无偏向性的方式以较宽的母离子窗口进行隔离并碎裂。为了分析SWATH-MS数据而开发的肽段为中心打分算法依赖于以先验的肽段色谱及质谱特性知识构建的谱图数据库及其肽段比对参数。本指南将介绍如何设计并建立一套SWATH-MS实验,如何进行质谱检测及如何进行数据分析。然后将介绍如何改进SWATH-MS数据采集流程,如何权衡相关参数设定及各类数据分析策略。
关键字:数据非依赖采集(data-independent acquisition); 质谱仪(mass spectrometry); 定量蛋白质组学(quantitative Proteomics); SWATH-MS; 系统生物学(systems biology)
引言
在过去的十年间,液相色谱联用串级质谱(LC-MS/MS)已经成为高通量蛋白质标准和蛋白质组学分析的首选技术 (Aebersold & Mann,2016)。该领域最近已经逐渐从解析几个样本中的蛋白质、肽段或翻译后修饰发展到数百个样本大规模、高质量且高重复性的定量组学。尤其在个性化医疗,生物标志物研究,药物筛选,基因相关研究或系统生物学研究中,大量不同状态、处理的个体样本需要进行生物学意义的挖掘。就这个目的来说,大规模定量数据的产出必须首先尽可能的可重复,完整而准确。近年来多个蛋白质组学检测技术就是为满足这样的需求而进行研发的。
其中的一个亮点就是本文介绍的由Gillet et al (2012)全部理论质谱谱峰顺序窗口采集技术(SWATHMS)。对于该技术的历史背景,我们已在前文介绍(表格1也进行了总结),不再赘叙。除了SWATH-MS外,还有一系列其他DIA方法和分析策略,我们在前文中已进行了简介,并且你在最近其他的一些综述里也能看到 (Chapman et al,2014; Bilbao et al,2015). 目前Sciex公司注册“SWATH”作为商标,应用于Q-TOF系统柜,Bignosys公司注册了 “Hyper Reaction Monitoring” (HRM) 作为Orbitrap系统上的应用商标。本文我们暂时用SWATH-MS来替代各类相似策略作为统称。而“DIA”则代表了这项数据采集技术本身。
何时选择SWATH-MS作为我的蛋白质组研究方法?
SWATH-MS的主要优势在于其可以在保证高稳定性和精确性的情况下定量数千个蛋白质。因此它非常适合于需要对大量样本进行蛋白质组或多肽组精确而可重复定量的研究。典型的应用研究诸如生物标志物研究 (Liu et al,2014; Muntel et al,2015; Kulkarni et al,2016; Ortea et al,2016),基因相关研究 (Liu et al,2015; Okada et al,2016; Williams et al,2016),药物/干扰组学研究 (Litichevskiy et al,2018; Tan et al,2017; Keam et al,2018) 或探索性基础研究 (Collins et al,2013; Lambert et al,2013; Parker et al,2015a; Schubert et al,2015b)。 SWATH-MS 同样特别适合用于快速短梯度检测研究 (Vowinckel et al,2018)。并能够单针检测就实现在复杂哺乳动物样本中约50%的蛋白质组覆盖率 (Bruderer et al,2017; Kelstrup et al,2018)。目前SWATH-MS相对于传统靶向蛋白质组研究(SRM or PRM)的劣势依然在于其灵敏度依然只有它们的1/3到1/10 (Gillet et al,2012;
Liu et al,2013; Schmidlin et al,2016)。因此,靶向数据采集技术依然是对低丰度蛋白和多肽高精度定量的最佳选择。另一个SWATH-MS相对于DDA技术的缺点在于其必须要先建立参考谱图数据库并需要对方法进行优化。在当前大规模的定量蛋白质组学研究中,有两项技术同样表现出色。第一个是基于DDA的非标记定量技术,其定量依据来自于母离子的信号强度或者谱图数量,且该方法可以结合色谱分级技术实现更高的蛋白覆盖度 (Lawrence et al,2015; Geyer et al,2016; Frejno et al,2017)。MS1定量技术实用化的一个主要技术进展是其数据分析工具对定量数据矩阵进行定性结果的跨样本匹配 (Prakash et al,2006; Mueller et al,2007; Cox et al,2014)。不过就算采用了该方法,DDA数据集的缺失值依然高于SWATH-MS模式,尤其在定量较低丰度蛋白质的时候 (Bruderer et al,2015,2017; Kelstrup et al,2018)。虽然直接比较MS1/DDA非标定量技术和SWATH-MS技术存在一定的困难,但也有多篇文献采用了同一个样品同时进行DDA和SWATH-MS检测的比较,并发现SWATH-MS在肽段检出量,定量可重复性上优于DDA(Bruderer et al,2015; Kelstrup et al,2018)。第二个流行的大规模定量蛋白质组策略是同位素标记,比如tandem mass tags(TMT) (Thompson et al,2003)或 isobaric tags for relative and absolute
quantitation (iTRAQ) (Unwin et al,2005)。标记定量通常需要对肽段进行预先的分级处理,然后每个分级产物分别进行DDA质谱检测(Chick et al,2016; Roumeliotis et al,2017)。在TMT10标记或11标记中(McAlister et al,2012),不同样品进行混合并同时上机检测,能够减少样品制备中的误差并提高样品间定量的一致性。这种方法也有一定的缺陷,比如由于共流出和共碎裂肽段导致的报告离子定量压缩效应,虽然也有一些优化的数据采集和分析方法进行了改进,但还是不可避免这个问题(Ting et al,2011; Savitski et al,2013; McAlister et al,2014; Ahrne et al,2016; O’Brien et al,2018; Sonnett et al,2018)。同位素标记适于中等规模的蛋白质组定量研究,但如果样品有数百个的话,其数据的完整性和批次效应会被放大导致标记批次间的一致性变得很差。目前暂时还没有直接在大规模样本水平比较同位素标记和SWATH-MS的文献。本文我们主要介绍SWATH-MS的实验流程建立,DIA质谱方法建立和数据分析。还会进一步介绍如何改进SWATH-MS实验,哪些参数是需要权衡的,以及有哪些不同的数据分析策略。
设计并建立一次SWATH-MS实验
如果你的SWATH-MS研究需要分析上百甚至上千个样品,那么需要特别想好如何获取可重复高质量数据,包括单个样品的检测时间以及是否要在多台仪器甚至多个不同型号仪器上同时采集同一类样本。目前令人振奋的是在不同实验室产生的同一套样本的SWATH-MS 数据具有较好的一致性(Collins et al,2017)。(译者注:当然最好避免在不同地点不同型号仪器上采集数据并进行整合,因为这类研究除了质谱外,其他处理和分离条件需要有个共同的严格流程才能实现较好的重复性)。
诸如脂类、聚合物或去垢剂在质谱中的累积是影响质谱表现的一个重要因素,且需要我们极力避免。SWATH-MS实验中污染物的影响尤其严重,我们能够观察到该类实验中由于污染导致的在SWATH-MS中相对于DDA或PRM更快速的灵敏度降低过程 (比如电荷效应和灵敏度问题)。一种解释是SWATH-MS模式下质谱引入了更多的碎片离子(>90%的数据采集时间),从而导致更多的污染物和其他杂质进入系统。因此,监测质谱性能并维持其相对稳定的表现状态是及其重要的。最近一些软件(如Biognosys的QUIC)已经致力于监控SWATH-MS的色谱质谱表现了(Rudnick et al,2010; Wang et al,2014; Bereman et al,2016;Chiva et al,2018)。当你在设计一套大规模非标记定量蛋白质组实验的时候,需要考虑实验设计的统计学因素,诸如分组规模,生物学及技术重复的变异度或可达到的灵敏及选择性 (Krzywinski & Altman,2014b)。为了保证下游数据分析合理的统计效力(Krzywinski & Altman,2013),我们建议重点考虑样本检测的随机化和区组设计 (Krzywinski & Altman,2014a),足够多的生物学和技术重复 (Blainey et al,2014)。
基于肽段中心的打分算法及谱图数据库依赖于先验的知识
SWATH-MS所基于的分析理念是关于目标肽段的色谱、质谱特性先验知识可以以靶向匹配的方式提取高度卷积的SWATH谱图中相应的肽段特征信息 (Gillet et al,2012)。这所需要的先验知识被称之为 “peptide query parameters” (PQPs)。需要注意的是 “prior先验”在这里的含义指的是PQP是需要在SWATH数据分析前先获取到的。PQP包含哪些信息呢?具体来讲:(i)已知蛋白所能检测到的相应肽段序列,(ii) 该肽段主要母离子的m/z值及其电荷分布,(iii) 当前仪器状态下该肽段最高丰度的4到6个碎片离子的m/z值,(iv) 当前仪器状态下肽段碎片预期的分布模式,如相对碎片离子强度和 v) 该肽段预期的保留时间及相应的碎片离子信号并与参照品进行归一化。PQP通常可以从谱图库中获取[也可以用色谱库 (Sharma et al,2014)] ,并以表格形式储存,如图1。
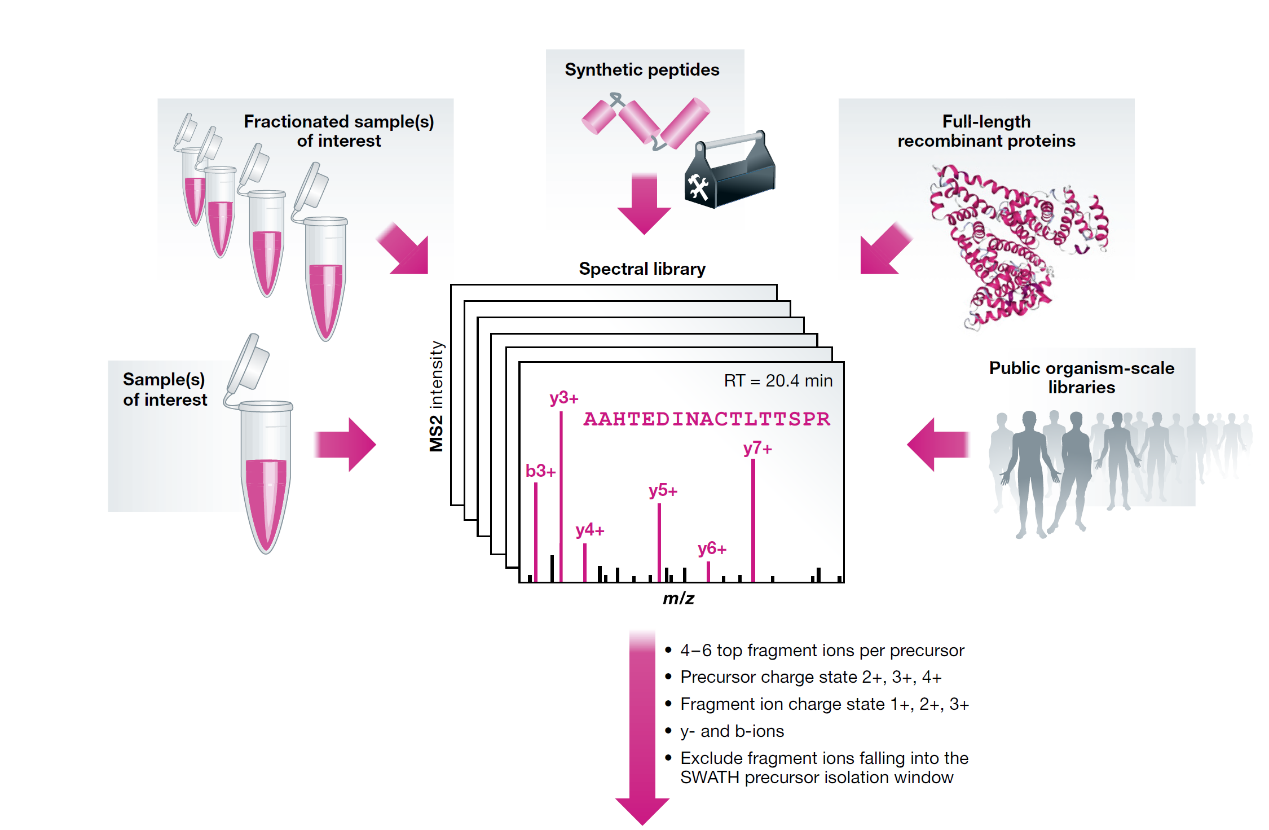
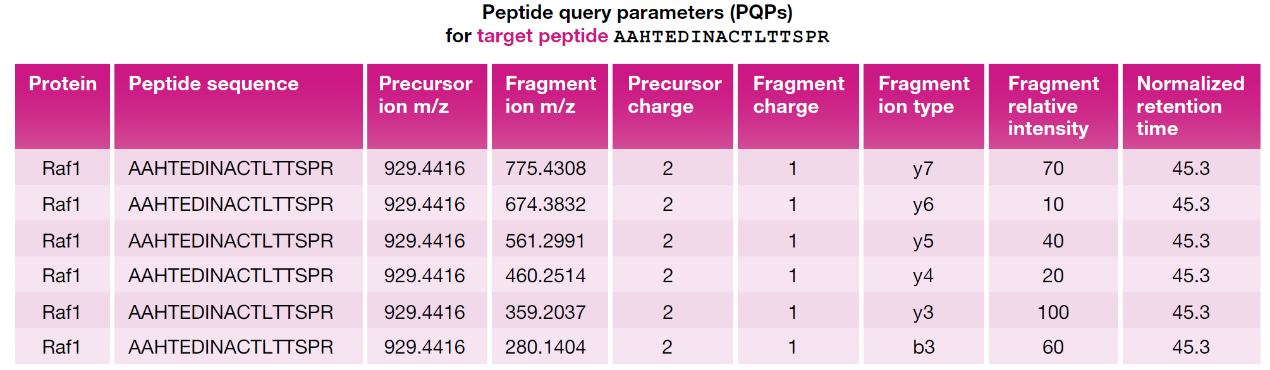
图1 PQP信息包含肽段的色谱和质谱特性,比如这边展示的肽段AAHTEDINACTLTTSPR。PQP信息可以来自于各类样本。当然最典型的就是该样本的DDA采集数据,所获取的信息有图1中的表格展示。从谱图库文件中的肽段定性结果出发,进行质控过滤后能提取到相对PQP信息。PQP包含了:对应的蛋白质,肽段序列,母离子m/z,碎片m/z,母离子和碎片的电荷数,碎裂离子类型,预期的相对碎片离子强度和归一化的保留时间(一般我们用iRT,相对于一系列参考肽段矫正而来的归一化时间)。
整合了谱图库建立的各个步骤并可以提取PQP信息的工具包括:Skyline (Egertson et al,2015),PeakView (SCIEX),Spectronaut X (Bruderer et al,2015) (Biognosys) 和Trans-Proteomic Pipeline (TPP) (Deutsch et al,2010)。还有一些工具可以预处理和转换[specL (Panse et al,2015),Fraggle/Tramler/Franklin (Teleman et al,2017)] 或拓展[SwathXtend (Wu et al,2016)] PQP信息库。MSPLIT-DIA工具可以用来优化和约束特别庞大的谱图库 (Wang et al,2015)。
如果你需要进一步了解如何从DDA数据建立谱图库和PQP,您可以参考最近的一篇方法文章(Schubert et al,2015a)(或者找我们了解SpectroNaut是如何快速建库的)。在接下来的章节中,我们将讨论建库用的信息类型和来源。
目前也有一类技术旨在不进行DDA数据采集而直接使用DIA数据,采用以谱图为中心的打分算法建库。当然这些方法尚处于起步发展阶段,最新的一些工具包括DIAUmpire (Tsou et al,2015),Group-DIA (Li et al,2015)和Spectronaut X (Biognosys),它们利用了目前最新仪器的卓越性能来改进DIA建库的固有缺陷。通常我们认为这些方法可以建立一套慢慢累积的谱图库,从而实现接近于不分级DDA数据的蛋白质组覆盖深度。
构建谱图库所需的代表性样本
我们需要确定用内源的样本肽段,合成标肽还是用重组全长蛋白来建库 (图1)。目前SWATH-MS相关研究主要采用研究对象构建的DDA谱图库。这些库通常来自于检测对象的分级DDA分析数据,分级可以帮助提高建库结果的灵敏度 (Rosenberger et al,2014; Zi et al,2014; Selevsek et al,2015)。这也是为何单针DDA分析的灵敏度和覆盖率一般都比SWATH-MS数据低的原因(Bruderer et al,2015; Kelstrup et al,2018)。因此,即使建库采用的是单针DDA谱图库,SWATH-MS数据也不可能覆盖整个数据库。反复进行单针DDA建库的策略有时候还挺受欢迎,当然主要因为是这个方法够简单;但是考虑到库的完整性和质量,我们还是应该考虑其他方法为佳。比如化学合成的标肽一直被认为是一个非常理想的先验知识的来源,尤其是在开发SRM方法的时候(Kuster et al,2005; Picotti et al,2010),甚至于合成和检测某些器官的全蛋白质组肽段也有报道 (Picotti et al,2013; Schubert et al,2013; Kusebauch et al,2016; Zolg et al,2017)。这个方法有如下优势: (i) 不管是否曾被检测到,所有蛋白都在收集在库里,(ii) 由于合成标肽的含量可控、组成确定,所以能够得到非常高质量的MS2谱图及其相应的PQP信息,及 (iii)谱图库的假阳性率几乎为0。在某些情况下,到底选蛋白的哪条肽段去合成依赖于经验指导。当然,目前几乎某几个物种在DDA模式下能够得到完全的蛋白质组覆盖,每个蛋白的酶切肽依然依赖于理论预测(Mallick et al,2007)。由于计算机预测的可靠性尚低于我们的预期 (Searle et al,2015),通过经验来选择肽段依然是必不可少的。最近的一些研究合成了蛋白质组规模的肽段(> 330,000人肽段),其中还包含PTM和常见序列变异(www.proteometools.org; Zolg et al,2017)。一种对这类策略比较有意义的衍生应用是用重组表达或转录组/翻译组系统的方法建立蛋白质全长序列。通过这种方法,每个蛋白最适合检测的肽段就能够通过经验找到(Stergachis et al,2011; Matsumoto et al,2017)。目前这些大规模合成肽段及蛋白的质谱数据还没有利用SWATH-MS分析,不过它们将是未来可利用的非常有用的资源。包含内源样品和合成标肽的混合库可以提高蛋白覆盖率,也是一种很有吸引能力的建库方法,目前已经在Mycobacterium tuberculosis这个物种的研究中得到利用 (Schubert et al,2015b)。
全面完整的谱图库和样品特异性的谱图库对比
以上讨论的一个常见推论就是到底是针对某个物种建立深度挖掘的DDA全库还是在每次研究的时候选择研究对象样本单独建库。这些库可以由样品本身或者合成蛋白质组来提供。现有的可供下载的器官水平的谱图库有S. pyogenes (Karlsson et al,2012),S. cerevisiae (Picotti et al,2013; Selevsek et al,2015),M. tuberculosis (Schubert et al,2013,2015b),H. sapiens (Rosenberger et al,2014; Kusebauch et al,2016; Zolg et al,2017),phyllosphere-colonizing bacteria (M. exotorquens,P. syringae 和S. melonis) (Muller et al,2016) 和E. coli (Ludwig et al,in preparation)。当然,今后类似的可供公共下载的不同物种来源用于SWATH-MS分析的先验知识库肯定会越来越多(Craig et al,2004; Martens et al,2005; Deutsch et al,2008; Picotti et al,2008; Sharma et al,2014; Whiteaker et al,2014; Wilhelm et al,2014; Zolg et al,2017)。目前SWATHAtlas (www.swathatlas.org)网站致力于收集可用于SWATH-MS分析且兼容大多数分析软件的谱图库。目前使用这些公共资源的一个重要前提是它们能否在不同实验室和不同仪器类型间通用。已有研究评估了不同仪器上建立先验的DDA谱图知识库的异同 (Toprak et al,2014)。尽管结论是最好在相同仪器平台上构建谱图库,但作者发现在肽段为中心分析算法的先验知识库构建中,不同仪器采用类似碎裂方式产生的碎片离子谱图具有相当的一致性 (比如 QqQ,QqTOF 和 Orbitrap HCD模式下的数据)(de Graaf et al,2011; Zolg et al,2017)。在利用了保留时间归一化(Norbeck et al,2005)和进一步采用索引保留时间(iRT) (Escher et al,2012)等算法后,不同实验室间的色谱数据也具有相当的可比性。这些方法的计算准确性在改变梯度时间和柱子长度后也是能够得到保证的;然而肽段的色谱流出顺序会受其他一些因素的显著改变,诸如流动相组成或柱温的改变,从而产生相当大的误差。最近一项跨全世界11个实验室的比较研究发现同一个器官的不同实验室来源谱图库可以有效的用于同一套SWATH-MS的分析(Collins et al,2017)。另一个在大规模谱图库资源使用过程中需要额外关注的问题是海量样本、海量肽段数据的错误率控制。在这类分析中,如果数据库中某一分级中的先验肽段信息完全不存在与研究对象中(可检出水平),那么其相应的假阳性率会非常之高,这个影响在大量样本的研究中会产生累积效应。如果SWATH结果在蛋白水平整合评估,其错误率会受到以上因素的显著影响(Reiter et al,2009; Rosenberger et al,2017a)。因此蛋白水平的假阳性率需要比肽段水平的更加受到关注,而其假阳性率的计算也需要基于全局样品计算而不是每个样品单独计算。 Rosenberger et al (2017a)最近发表的研究建立了一种从发现式蛋白质组学中总结而来的统计方法,能够较好的控制SWATH-MS实验中肽段和蛋白水平的错误率。综上,采用基于器官特异性的现成数据库能够大大降低建库的工作量,但这样会因为海量数据的多重检验及矫正统计负担,导致丧失一定的统计学效能,换言之就是结果不一定尽如人意。折衷方法可以考虑并不直接检索谱图全库而是仅仅提取库中和研究对象或者相关生物学功能比较密切的一部分蛋白作为数据库。
由靶向到发现式的SWATH-MS技术,到底应该关注哪些和多少肽段呢?
在最初的SWATH-MS操作中,其主要目的是用来拓展SRM技术的检测范围的 (Gillet et al,2012)。因此其首先是针对某一特定生物学问题相关的蛋白质来定量。但很快人们发现SWATH-MS可以利用谱图库提取复杂样品中大量肽段的离子色谱信息。随着时间推移,一次完整SWATH-MS研究可以获取肽段定量信息显著增长,在发展了合适的错误率控制算法后(Rosenberger et al,2017a),现在可以利用器官特异的谱图库及打分算法分析数十到十万条肽段,从而是该技术从服务于靶向蛋白质组演化成了发现式蛋白质组技术(Bruderer et al,2017)。
到底是选择一系列感兴趣的目标蛋白还是全蛋白质组来分析取决于你的实验假设和目的,比如你检测到的这些蛋白质是否能够有效解释相关的生物学问题。如果一个生物学假设能够由一些选定的目标蛋白质的量变来解释,那么增加检测范围反而会给实验数据的统计带来额外的负担。而如果生物学假设无法用一小群特定蛋白的变化来解释的话,那么利用谱图库来检测尽可能多的肽段则可能为你提供足够丰富的结果信息,其带来的额外统计负担则可以忽略。
由于分子量、保留时间相似的污染肽段干扰,目标肽段的分析往往会受到假阳性信号的影响,这也是基于肽段中心的打分算法需要考虑的问题。比如序列高度相似的不同肽段、同一肽段发生修饰的分子量差异和肽段骨架上氨基酸分子量差异相同。最近一项基于DDA数据开放式修饰搜索的研究发现修饰对此的影像相对较小 (Kong et al,2017)。开放式修饰搜索给出的非常见修饰肽相对于非修饰肽的比例也就大约1%。这也意味着修饰肽对SWATH-MS结果的假阳性率影像也微乎其微,不过后续新算法的开发还是得关注下相似肽匹配的可能性(Rost et al,2012)。目前的一些数据分析方法开发文献也开始考虑“peptidoforms”对SWATH数据的影像(Rosenberger et al,2017b)。
综上,碎片离子数据的质谱、色谱高完整性优势使得DIA技术为多种数据分析需求提供了技术解决方案。其技术原理类似于SRM,因此也能够灵活地检测全蛋白质组或目标肽段。
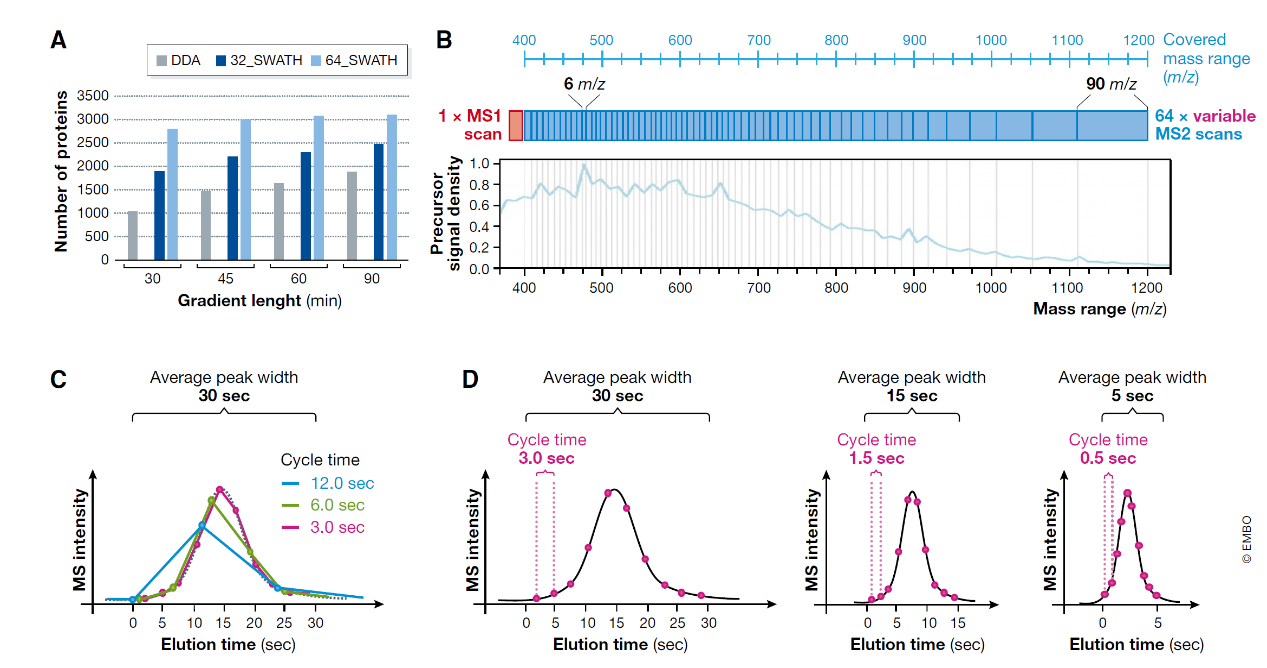
图2 确定一个最佳的SWATH-MS折衷方法。(A) HEK细胞裂解液在DDA模式(灰色)和SWATH 32个固定窗口(深蓝)及SWATH 64个可变窗口(浅蓝)在不同色谱梯度长度下定性数量的比较,质谱采用Q-TOF系统。(B)为了改进母离子的选择性,可变母离子窗口可以根据各个m/z范围内的谱峰密度进行调整。(C)如果要精准的进行谱峰重构,一般每个母离子需要采集10个数据点。对于一个峰宽30秒,循环时间3秒的谱峰,即能采集到10个数据点(红色)从而能够非常精确地重绘实际谱峰(灰色虚线)。如果循环时间更长,比如6秒(绿色线)或12秒(蓝色线)则会导致谱峰形状失真。(D) 如果峰宽从30秒(左)缩短到15秒(中)甚至5秒(右),为了构建足够的10个数据点所需的循环时间对应于3、1.5和0.5秒。
进行一次SWATH-MS检测
设置SWATH-MS的色谱方法
SWATH-MS的液相色谱设置和常规靶向及DDA实验并无太大区别。一般采用0.5-2ug肽段混合物载入nano-HPLC系统,并用15–50厘米长的反相色谱柱(比如3或1.8um的C18填料),在300 nl/min的流速下采用逐步增加有机相配比的方法洗脱。另外~5ul/min的microflow HPLC 系统也可以兼容SWATH-MS,就是灵敏度会损失3-5倍且需要进样3-5倍量的肽段样品 (Bruderer et al,2017; Vowinckel et al,2018)。
基于肽段中心分析SWATH-MS多窗口采集的方法依赖于采集足够多的数据点(典型值就是一个肽段10个点),来构建足够精确地色谱峰 (图2C)。现在SWATH-MS需要用2-4秒循环时间来覆盖整个母离子采集范围,同时每个谱峰需要采集到足够的数据点,因此平均色谱峰宽不能低于20-40秒。单窗口的DIA方法就截然不同,它只在单个MS1和MS2间进行采集循环,因此可以容纳2-3倍的高性能LC产生的色谱峰宽[比如AIF (Geiger et al,2010)或MSE (Silva et al,2006)]。随着质谱仪的扫描速度逐渐提升,更窄的色谱峰能够与多窗口SWATH-MS方法更好地兼容。另一个有趣的事情是SWAHT-MS能够很好地和短梯度HPLC兼容。DDA模式下梯度越短,采集到的MS2谱图及鉴定到的肽段随之成比例下降,而SWATH-MS中MS2采集在较短的梯度下并不会造成肽段鉴定的大量损失(图2A)。SWATH-MS模式下缩短梯度而导致的肽段数目变少可能是和母离子共流出增多及同时发生的离子信号抑制和共碎裂效应,而不是来不及采样。当然随着质谱仪器性能进一步的提升,SWATH-MS必将在较短的梯度实现更好的选择性和鉴定率,从而实现更高的检测通量。
综上,我们建议采用2小时nano-HPLC梯度来采集高质量的谱图库(Schubert et al,2015a),同时为了实现更高通量,SWATH-MS采集的时候也可以考虑缩短梯度到比如30-60分钟,其选择性和蛋白质组覆盖度也不会降低的太多。
建立SWATH-MS数据采集方法
确定一个最佳的折衷方法
SWATH-MS采集过程最优化的母离子(MS1) 和碎片离子(MS2)扫描参数取决于一系列因素,如分析对象预期的分子量范围,样品复杂度,质谱仪器本身的分辨率和扫描速度,LC设置及其色谱峰宽,还有就是你预期的灵敏度和选择性。实践中我们需要关注的参数如下:(i)需要覆盖的母离子m/z范围,(ii)母离子隔离窗口的宽度,(iii)碎片和母离子累积/扫描时间和分辨率,(iv) 色谱循环时间和(v)每个样品注入量。在以下章节中,我们将详细讨论每个参数的影响。本文作者在Q-TOF类型仪器上的经验比较丰富,所以我们的建议都基于各自的使用、优化和仿真经验(Rost et al,2012)。不过,随着Orbitrap仪器目前在SWATH-MS分析中越来越常用,我们同样会提及该平台上成功使用并发表的相关文献。采集窗口的详细举例参见表格EV1,包括Q-TOF和Q-Orbitrap仪器。这些参数可以作为你产出高质量SWATH-MS数据的一个起点;当然后续基于此的优化肯定可以得到更多的可定量蛋白质和肽段。需要最后指出的是,Q-TOF和Q-Orbitrap仪器上SWATH-MS的采集参数以后肯定还有很大的优化空间。
覆盖的母离子m/z范围
SWATH-MS检测覆盖的母离子m/z范围取决于一系列母离子隔离窗口合并后覆盖的区域。理想情况下,质量范围当然是尽可能的覆盖所有肽段出峰的m/z。检测对象如果落在范围外自然是没法检测及定量的。所以如果你的目的是检测蛋白质组中尽可能多的部分并且采用了胰酶酶切,那么肽段最丰富的m/z范围是400–1,200 m/z (Gillet et al 2012)。在部分仪器上,该范围也可以缩小到500–900 m/z (Egertson et al,2015) ,同时不会损失太多定量蛋白。如果你用了其他酶或者并不是分析肽段(比如代谢物),那么母离子覆盖范围自然得相应调整。Q-TOF和Q-Orbitrap仪器较为常用的范围我们也详细列在了表EV1里了。
母离子隔离窗口设置
母离子隔离窗口定义了每个MS2扫描所共采集和共碎裂的母离子质量范围。因此,隔离窗口宽度直接影响了选择性和检测的动态范围。该参数应该也是在不同实践方法中区别最大的,从800 m/z一个窗口到数百个2m/z窗口不等。
虽然较宽的隔离窗口能够让质谱仪循环扫描的更快,但同时也会共碎裂更多的肽段母离子,从而导致MS2变得更加卷积,以及灵敏度也随之变低。相反的,较窄的隔离窗口能够得到较少的共碎裂母离子和信号干扰,但其他参数比如能够覆盖的母离子范围或每个色谱峰采集到的数据点可能就不够了。
有一点需要注意的是每个母离子隔离窗口的大小都是可以单独设置的。采用可变窗口能够获得更好的鉴定数或者母离子在各个窗口中的强度分布会更均匀(Zhang et al,2015)(图2B)。这种优化可变窗口的方式可以在每个样品或者同一个实验多个样品中分别设置。目前也有了一些基于经验数据的自动设置窗口软件(比如SCIEX的“SWATH Variable Window Calculator”)。经过实践发现,优化窗口宽度并不会显著影响肽段数量或者定量结果。为了证明这一论断,我们在TOF仪器上采用了一套“通用的64个可变窗口”(表EV1)的采集方式,结果发现其可以通用的分析复杂性从人器官到细菌等不同的样品。这种母离子隔离策略可以从人细胞系中提取到约50000个酶切母离子信号,从5.9 m/z宽度[472.4–478.3]范围的最高密度信号窗口,到90.9 m/z宽度[1,109.6–1,200.5]范围的低密度信号窗口。从原来的32个固定窗口到64个可变窗口,我们发现定性和定量上都得到了提升(Navarro et al,2016)。Bruderer et al在Q-Exactive 优化得到19可变窗口设置(Bruderer et al,2015) ,而在Q-Exactive HF上设为24个可变窗口(Bruderer et al,2017)。Kelstrup et al(Kelstrup et al,2018)发表了Q-Exactive HF-X上用的72个可变窗口方法 (表EV1)。
最近,一些新的DIA采集方法也使用了一些不太一样隔离窗口设置 (Egertson et al,2013;Moseley et al,2018)。肽段隔离过程中不同的干扰因素在这些方法中起到的左右又各自不同,因此都需要特殊的去卷积工具进行去卷积、后处理。
碎片和母离子累积时间和解离能量
对于Q-TOF仪器来说,质谱采集或累积时间决定了质谱仪对某个特定MS扫描能够积攒多少离子信号。累积时间越长,则信噪比越好。但是,较长的累积时间将影响质谱的循环采集时间。因此,累积时间必须和母离子隔离窗口的大小相适配(图2C)。对于Orbitrap质谱来说,质谱采集时间分为两个平行的时间相隔过程。首先,“注入时间”用来收集离子阱中有自动获取控制(AGC)参数控制的所需数量的离子。其次,“扫描时间”是Orbitrap所需近路质谱谱峰的时间,其取决于分辨率。在新一代Orbitrap质谱上(Q-Exactive系列以及Fusion/Lumos),这两个过程是平行进行的,因此更耗时的那一步决定了循环时间。典型的最大离子注入时间由所需的扫描时间来确定。近期的相关文献报告采用19个(Bruderer et al,2015)(Q-Exactive)和24个可变窗口(Bruderer et al,2017) (Q-Exactive HF) ,分辨率30000 (Full Width Half Maximum) 或者70个窗口(Kelstrup et al,2018) (Q-Exactive HF-X) 固定9m/z窗口,分辨率15000 (表 EV1)。随着仪器性能的快速提升,将来一定会有更优化的参数。
综上,Q-TOF质谱联用nano-LC 系统,平均色谱峰宽约30秒,采用64个可变窗口的时候,我们建议MS2累积时间约50毫秒。其完整循环时间约为3.3秒。而Q-Exactive HF采用30000分辨率,24个可变窗口 (Bruderer et al,2017),可以得到相似的循环时间。当然,由于加入了AGC,扫描时间和工作周期这些影响因素,Orbitrap仪器需要进一步单独优化。我们还建议在Q-TOF仪器的每个循环MS2扫描前加入一个250毫秒的MS1扫描。Orbitrap仪器同样建议加入一个或数个MS1扫描。来自于MS1的信息可以在一些最新的DIA分析软件中得到充分利用,比如用来辅助确定肽段的母离子分子量或者辅助共流出碎片离子的去卷积。
色谱循环时间
在SWATH-MS中,“循环时间cycle time”,或叫“工作周期duty cycle”或者“采样率sampling rate”对应的是MS1和MS2扫描的累积时间总和(及加上仪器内部开销时间,可忽略)。因此,循环时间定义了肽段的同一个离子在色谱流出时如何被采集的。循环时间对任何基于色谱信号定量的方法都是至关重要的。一般来说,构建色谱谱峰峰型需要10个采样数据点来进行精确定量 (图2C) (Matthews & Hayes,1976; Lange et al,2008)。不过采集到10个数据点的平均色谱峰宽各自不同,其取决于色谱设置。在一个平均峰宽(基线处)大约为30秒左右(3 um C18填料的nano-HPLC,梯度时间1-2小时的典型值)的实验中,循环时间设为3秒比较合适。当然,如果你使用了一个更高分辨率的色谱系统,平均肽段流出峰宽可能会窄到5秒的话,循环时间可以较少到0.5秒(图2D)。
每个样品的注入次数
对于数百个样品规模的蛋白质组学研究来说,单针进样是保证通量和定量一致性的最佳选择。不过小规模实验的话,每个样品多针进样(N),每针覆盖不同的m/z范围则是一个挺有吸引力的用以增加选择和灵敏度的替代方案(Panchaud et al,2009; Ting et al,2017)。这样可以缩小母离子隔离窗口宽度N倍,总而保证覆盖一个较大的母离子m/z范围。(译者注:Spectronaut也支持分级DIA数据分析)
设置碰撞能量
在DDA,SRM和PRM采集方式中,碰撞能力可以对母离子进行设置(基于母离子m/z和电荷数),或者更可以针对特定的transition设置 (SRM),然而在SWATH-MS中只能对每个隔离窗口设置碰撞能量。换句话说,如果两个肽段有不同的电荷数且位于同一个隔离窗口内,他们会在相同碰撞能量下共碎裂,这样看着似乎不那么合适。不过这样欠妥的肽段碎裂在某种程度上由谱图库的信息来弥补。可以在谱图库中提取DDA碎片离子,并转换成电荷数无关的碰撞能量公式,比如碰撞能量随母离子m/z而变化但和电荷数无关。需要注意的是这个策略会导致DDA分析结果在高电荷数肽段的鉴定数量下降。这种损失也可以部分通过在一个SWATH窗口中斜升碰撞能量来弥补,比如同时在DDA和SWATH模式MS2累积时间中,从-15渐进到+15 V。
综上,在Q-TOF仪器中,我们建议在DDA谱图库构建时,碰撞能量的设置和m/z为2电荷的母离子成比例,并且在SWATH-MS中,在隔离窗口中心的理论2电荷母离子也设为相同能量。我们还建议系统性的评估不同仪器及公共数据库的碎片相似度。目前也有软件支持这样的比较分析(Toprak et al,2014)。如果发现样品谱图库和SWATH采集的碎裂模式差异较大,我们建议最好优化碰撞能量以达到最大的碎裂相似度。这样能够保证不同肽段的匹配参数在不同仪器和实验室间能轻松对接。
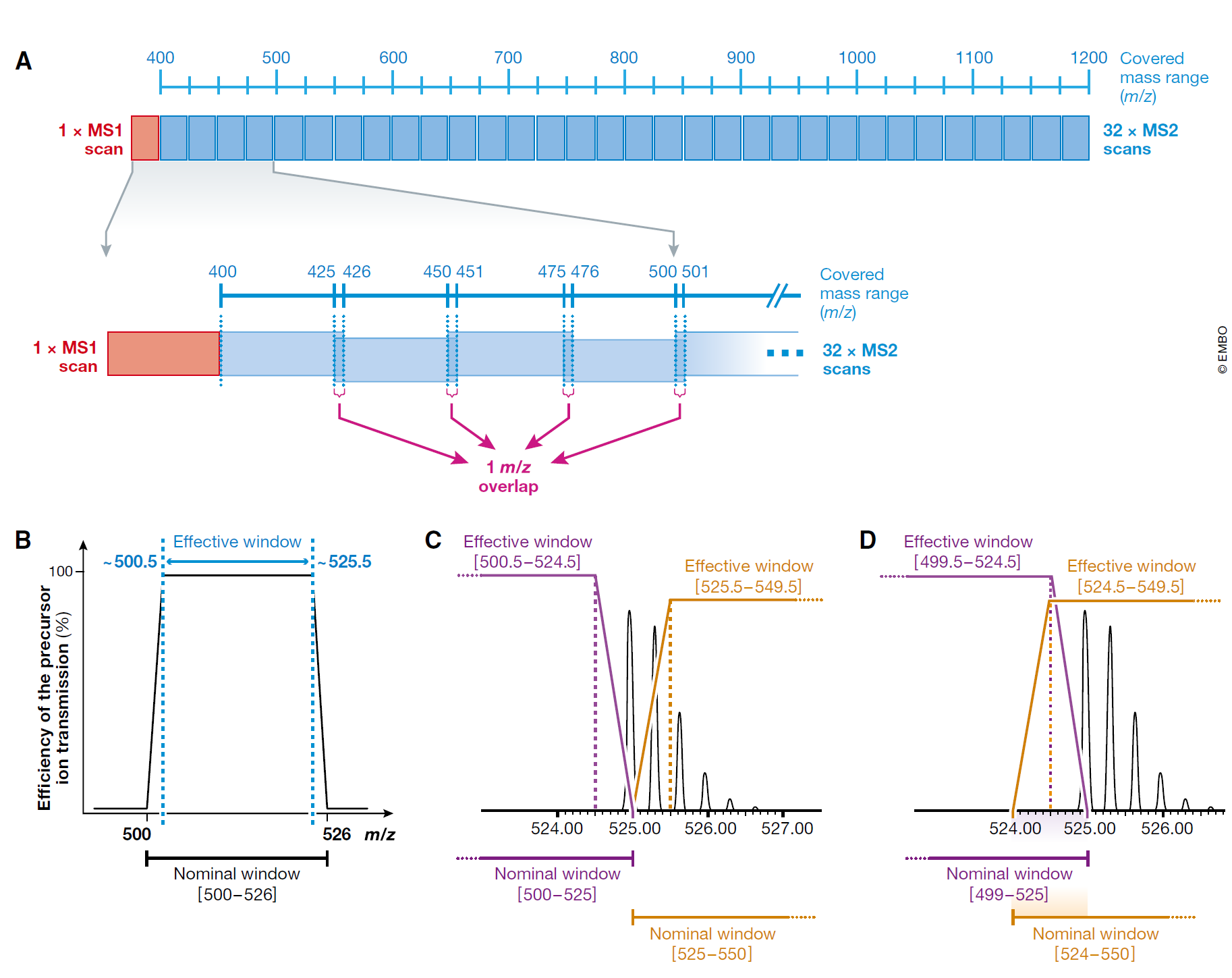
图3 采用轻微重叠的母离子隔离窗口的原理解释。(A) 在早期SWATH-MS方法优化中,相邻的SWATH窗口会有1 m/z的重叠以避免四级杆选择离子时在隔离窗口边缘产生的传输效率损失问题 (B) 为了降低母离子同位素被相邻窗口隔开的效应(C and D)。(B)图解离子传输效率在四级杆质谱中以 [500–526] m/z范围为例的衰减情况。(C) 二电荷肽段MLSYPITIGSLLHK (m/z = 524.965)理论同位素分布。如果不采用窗口重叠的话,[(500–525)和(525–550)]间的同位素信号会大幅衰减且被分隔。就算窗口边缘没有母离子(比如525.1),离子衰减依然存在,同位素分布信号也会失真。(D) 在窗口重叠的情况下[(499–525)和(524–550)],大多数母离子同位素都会被有效传输[如(499.5–524.5) 和(524.5–549.5)]。
相邻的母离子隔离窗口是否需要重叠?
在最初的SWATH-MS文章中,我们建议相邻的SWATH窗口有1m/z的母离子隔离窗口重叠(图3A和表EV1) (Gillet et al,2012)。这么做有两个理由:第一,四级杆中的母离子传输在整个隔离窗口分子量范围内不都是100%的效率,而在边缘处会减低(图3B)。因此,一个微小的窗口边缘重叠会补偿这样的损失。第二,重叠窗口保证了母离子同位素峰的最大完整性,避免了母离子在窗口边缘被分割后无法重构完整的同位素分布的问题(比较下图3C 和D)。重要的是,除非整个母离子同位素分布被隔离,碎片离子都会因此而受干扰,从而使得其数据匹配打分降低(见后续章节“自动谱峰分组打分”)。至今为止的SWATH-MS相关采用1m/z重叠窗口的文献也不都是因为以上如此极端的原因而这么做的,同样的,更大一些的隔离窗口重叠在将来也是值得考虑的策略。就算母离子同位素分布在两个相邻窗口间会被分隔(见图3C),该肽段的非标定量差异值在不同样品检测的时候也是正确的,只要你设置的窗口保持不变(Egertson et al,2015)。当然,如果你在类似的实验中采用SILAC类型的实验设计从而导致轻重离子对可能其中之一被分隔在两个窗口内,那么轻重离子比值可能会有失真。另一个可能降低该效应的方法是将隔离窗口的质量边缘设置为极少可能出现肽段的数值Egertson et al (2013)。这些理论上最优的窗口设置可以通过Skyline软件轻松设置(Egertson et al,2015)。尽管这样做这样并不能解决窗口边缘的信号损失问题,也不能防止母离子分隔在不同窗口,它可以保证单同位素峰不会正好落在窗口边缘。
综上,Gillet et al(2012) 提出的1 m/z重叠窗口的方法因为检测窗口被略微放大,会略微降低选择性。但它带来的好处是巨大的,可以将肽段信号在隔离窗口边缘的损失效应降低,得到更完整的同位素峰分布。当然,如果你不想用这个方法,那就最好用Egertson et al (2013)提出的优化窗口设置的方法了。
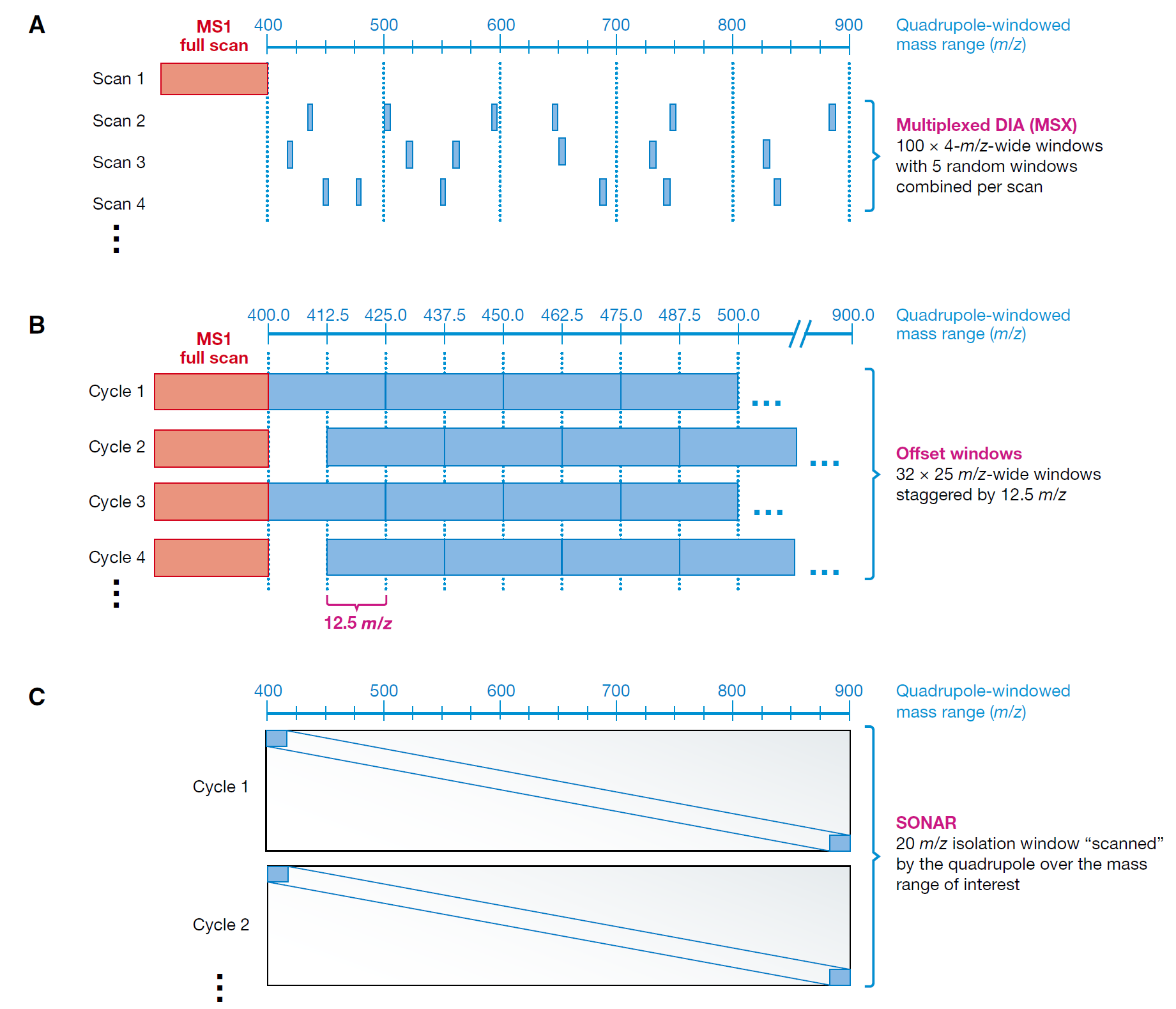
图4. 改善母离子选择性的一些新DIA采集方式。(A) Multiplexed DIA (MSX) (Egertson et al, 2013) 通过设置非连续的母离子窗口来改善选择性。(B) 在offset-windowed DIA方法中,母离子隔离窗口边界在连续扫描间加入一个偏移量,比如25 m/z的隔离窗口在每次循环间边界进行12.5m/z的偏移。(C)“scanning quadrupole”隔离方法,也称之为SONAR (Moseley et al, 2018),质谱仪连续无级扫描整个感兴趣的母离子分子量范围,比如500到900 m/z,使用200 × 20 m/z宽的窗口,2 m/z增量扫描。
改进母离子选择性的DIA优化采集方式
SWATH-MS的选择性是由母离子隔离窗口宽度直接决定的。而选择性则直接影响了最终达到的动态范围和SWATH-MS检测能实现的灵敏度。最近也有一些更新的SWATH-MS检测技术能够改善选择性(图4)。
Multiplexed DIA (MSX)
2013年,MacCoss课题组报道了MSX技术(Egertson et al,2013),一种多通道DIA方法。不像SWATH方法采用20m/z宽度来选择母离子,MSX采用每个循环中非连续的采集不同的隔离窗口的方式(比如5个非连续的4m/z宽的隔离窗口图4A)。在MSX方法中,每张谱图总的绝对选择性和SWATH-MS的20 m/z是一样的。然而在MSX中,一个特定肽段母离子会在每个循环中被随机共碎裂到不同离子群中,使得其受到的检测干扰在流出过程中各不相同。在其最新的改进方法中,MSX需要质谱仪器能够多通道的诱捕离子能力(这样排除了Q-TOF类仪器)
隔离窗口偏移技术
在隔离窗口偏移技术中,母离子的隔离窗口边界在连续循环间被反复正向和反向大幅偏移 (比如25 m/z的隔离窗口,偏移量12.5 m/z; 图4B)。和MSX类似,偏移采集技术产生了不一样的母离子隔离和共碎裂谱图。因此,来自同一群离子的共碎裂干扰可以通过该方法来进行计算消除。该方法改进了选择性并降低了定量限制,且并不需要额外牺牲循环时间、累积时间和分子量覆盖范围或分辨率。(https://skyline.gs.washington.edu/labkey/files/home/software/Skyline/2013-ASMS-Overlapped-DIA.pdf)。但是,该方法需要在数据去卷积前先用自动解析算法来预检原始数据或者原始碎片离子的提取。不像MSX,该方法也可以在非离子阱类仪器如Q-TOF上使用。
Scanning quadrupole isolation
最近的一个SWATH-MS的新技术叫做“SONAR” (Waters) ,由Moseley et al (2018)开发。 不再进行渐进式的隔离窗口循环扫描,该技术中四级杆会进行无级“scans”,比如,以24m/z宽度的隔离窗口在整个感兴趣的母离子质量范围内无级循环扫描 (图4C)。换句话说,在传统SWATH-MS方法中,某个特定的母离子只会出现在一个隔离窗口中,而在 “scanning windows”中,我们可以看到随着母离子同位素反复进入和离开扫描窗口而产生的碎片离子信号的出现和消失。这就在SWATH-MS数据中所包含的保留时间,质量数和强度维度外又增加了第四种维度。该方法需要大量改动已有的数据分析工具,但额外增加的第四维度信息同样可以增加数据的选择性。
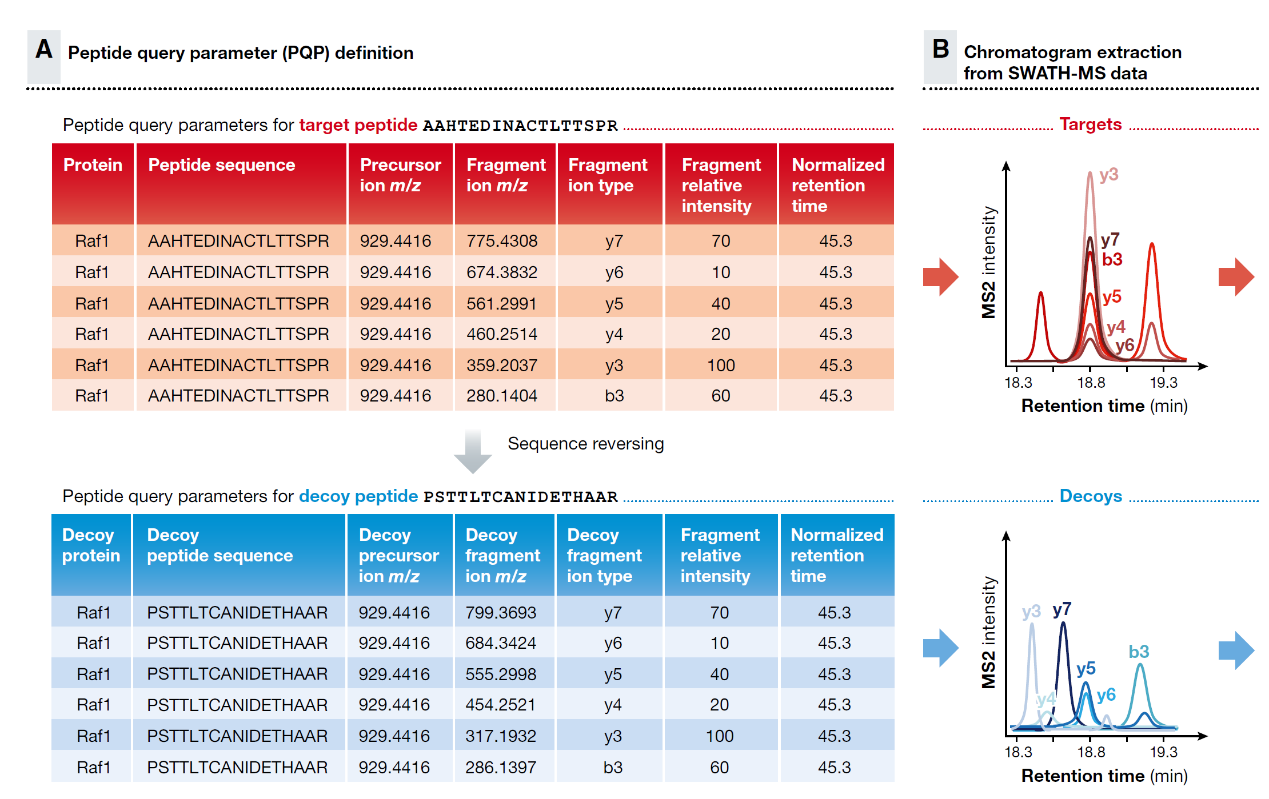
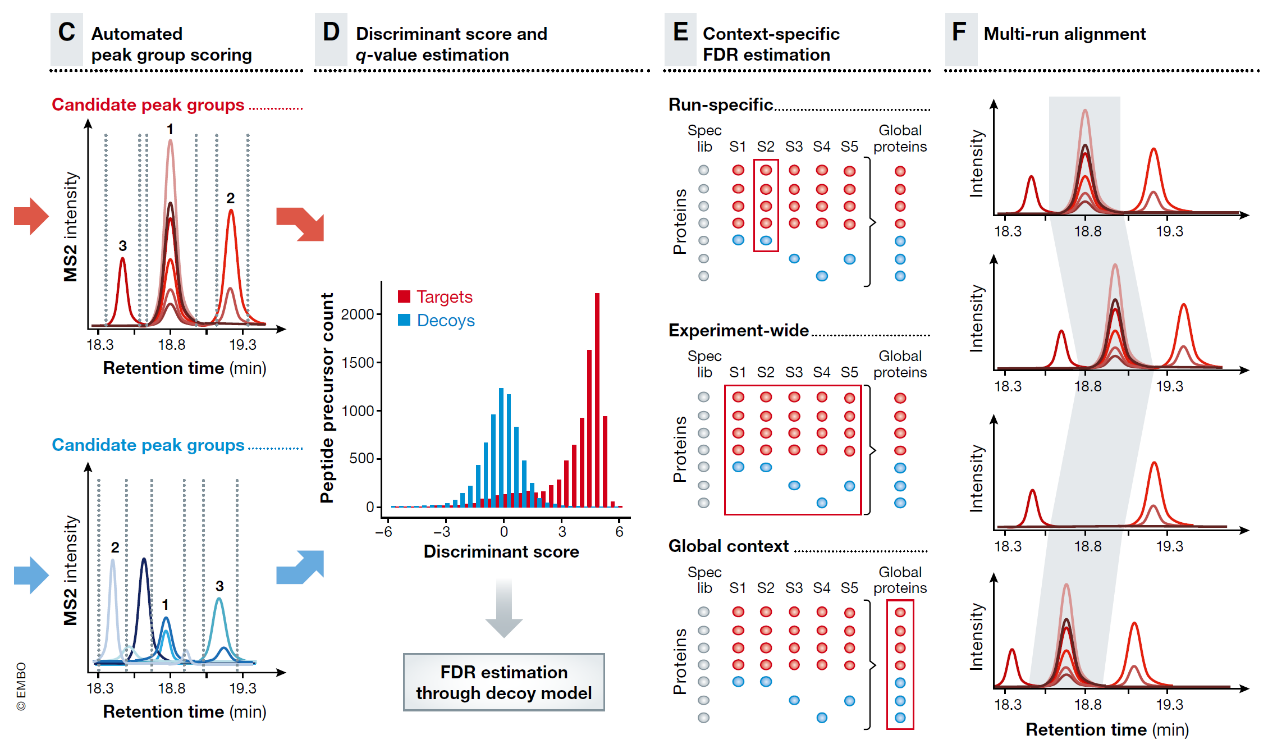
图5. SWATH-MS数据基于肽段中心打分算法的原理。(A) 肽段中心打分算法首先需要一系列肽段查询参数(PQP),包括目标肽的保留时间,母离子质量数,碎片离子质量数和碎片离子强度信息(红色表格)。Decoy肽段的PQP信息在分析时同样也需要对应产生,比如反转目标肽段的氨基酸序列同时保持末端氨基酸不变(蓝色表格)。Decoy肽段被用于评估肽段的定性结果是否是随机匹配的可能性。(B) 目标肽段和Decoy肽段在SWATH-MS2谱图中连续采集构成的PQP生成的提取离子流图(XICs)。这使得经过转换和精简的数据结构和靶向蛋白质组(SRM或PRM)很相似。(C) 碎片离子色谱图是根据其对应的肽段关联性进行组合,并选定有确定保留时间范围的 “peak groups谱峰组”。(D)同时针对目标和decoy谱峰计算基于色谱和质谱峰信息的得分分布,并基于半监督学习算法组合成判别得分(Discriminant score)。一系列检测到的肽段的错误发现率(FDR)可以通过目标和decoy肽段的得分分布进行统计建模来评估。(E)对于大规模SWATH-MS分析,错误比例的控制不仅仅需要针对肽段水平,更需要扩展到蛋白水平进行评估。进一步来说,大规模实验包含了大量的样品,每个单独样品进行错误比例控制并不足够严谨,最好在“整个实验规模”的进行控制。“全局”环境考虑的是基于所有样品的检测到的谱峰组,肽段或者组合的蛋白。(F)在多个样品间利用色谱时间一致性进行谱峰对齐并在样品间转移定性结果可以帮助矫正或增强谱峰检测的可信度。
SWATH-MS数据的肽段中心打分
过去20年出现过多种多样的DIA数据分析策略。这些策略总的来说可以分为两大类,基于谱峰为中心或基于肽段为中心的数据查询方法(Ting et al,2015)。本教程我们重点关注依赖色谱信息的肽段为中心的分析流程,我们将在下面着重讨论其每步算法并在图5插图说明。对于其他分析策略,可以参考以下综述(Chapman et al,2014; Bilbao et al,2015)。已有不少软件包含图5中的绝大多数分析步骤。比如有OpenSWATH (Rost et al,2014) (OpenMS的组件,Rost et al,2016b; Sturm et al,2008),Skyline (MacLean et al,2010),PeakView (SCIEX) 和Spectronaut (Bruderer et al,2015) (Biognosys)。最近也有文章专门用一套实际数据对以上这些肽段中心算法软件以及谱图中心算法软件DIAUmpire(Tsou et al,2015)进行了对比(Navarro et al,2016),并且发现它们之间的定性、定量一致性还是非常好的。(译者注,其中表现最佳的是Spectronaut)
(PQP) 肽段查询参数的定义
肽段中心分析的第一步工作就是要定义PQP。最近一些新算法可以不依赖于凭经验获取的PQP (Tsou et al,2015; Ting et al,2017),因此可以不再使用构建先验知识的谱图库了,由实验数据里的碎片离子谱峰构建的PQP同样可以提供较高的灵敏度,尤其是采用较大的母离子隔离窗口(>5 m/z)的时候。取决于样品的复杂性,一般都能给每个肽段找到多个高质量的谱峰组(peak group)。我们采用和目标肽性质,如色谱和质谱参数空间类似但不会存在于样品中的decoy肽段来定量描述这些谱峰组的出现频率。Decoy PQP由目标肽段氨基酸序列反转或随机组合模拟而来(图5A)。因为Decoy肽段可以用于评估共流出的色谱峰质谱碎片离子的随机效应,对于控制SWATH-MS数据中肽段检测和蛋白组装的错误比例来说至关重要(Rost et al,2014)。由于在肽段匹配时采用6个共流出碎片离子和采用3个共流出碎片离子的得分分布并不同,我们建议对目标肽段和decoy肽段进行谱峰打分时采用相同的碎片离子来避免打分偏差。
色谱峰提取
肽段中心数据分析策略的下一步就是采用定义的PQP来提取目标肽段的母离子及碎片离子的色谱峰信息。随着肽段从色谱柱流出,它对应的碎片离子强度会随之按照高斯分布的肽段流出曲线同步变化,从而构成“peak group谱峰组”的色谱峰用于评估肽段检测结果及其定量值(图5B)。目标肽和decoy肽段是采用相同的方式来提取碎片离子色谱峰的。实际上这步提取步骤转换并精简了SWATH-MS2谱图,并使之非常近似于SRM或PRM数据。为了充分改进肽段中心的SWATH-MS数据分析的选择性,色谱峰提取不会在整个色谱梯度上提取而是会基于目标肽的预期流出时间附近的保留时间窗口中进行(和scheduled SRM方法类似)。为了尽可能调整和预先构建的谱图的流出时间变形及限制保留时间提取窗口的大小,我们会采用检测一系列内源性(Parker et al,2015b; Bruderer et al,2016) 或掺入合成的肽段(Escher et al,2012)。这些参考肽的特征流出时间作为锚点来进行保留时间归一化(例如iRT方法; Escher et al,2012) ,然后样品中的肽段保留时间可以据此对齐。自动采用归一化的索引保留时间对齐功能在各类SWATH-MS数据分析软件中都已采用 (比如 OpenSWATH,Skyline,Peak-View和Spectronaut)。质量容差或提取离子峰宽也同样直接影响了色谱峰信号的选择性和获取的定性结果打分及定量计算准确性。一般来说,提取峰宽是质谱峰的半峰宽的话能够在最大化离子信号获取和保证可靠地选择性间获得一个很好地平衡。实际上,我们建议在MS2分辨率为15000的时候采用提取峰宽约50ppm或更低。
MS2数据保存为profile或者centroid都可以进行色谱峰提取。Centroid的MS2数据文件明显小于profile,而且分析结果定性数量和定量准确性上和profile几乎没有区别(Navarro et al,2016)。Centroid数据的提取窗口宽度可能没法像同一个数据profile模式优化的那么好,因为profile的谱峰可以进一步进行质量数错误矫正。不过数据centroid算法对数据分析影响很大。我们发现这对质量精度、信噪比和相对碎片离子强度影响很大(比如,centroid是如何处理峰高和峰面积的) (Toprak et al,2014)。由于肽段共碎裂谱峰的高度复杂性和高度互相交盖,SWATH-MS数据的centroid难度依然很高。这种和算法相关的人为影响会导致碎片离子谱峰组打分和肽段检测的统计结果。因此我们建议遵从仪器厂商和算法开发者的建议来选择合适的centroid算法。
最后,如果SWATH-MS数据在循环时间内也采集了MS1数据,母离子的离子流也能够提取并打分计入碎片离子谱峰组结果,用以提高肽段鉴定置信度。
自动谱峰分组打分
在数据分析流程的下一个步骤里,一个或多个潜在的色谱谱峰组的左右边界由专门算法来定义,并对每个候选的肽段包括目标(target)序列和decoy序列单独进行打分(图5C)。在打分过程中计算得到一系列不同的独立得分 (Reiter et al,2011)。然后被分组为五个类别:(i) 得分和色谱表现相关,(ii) 得分反映了检测到数据和谱图库的相关性,(iii) 得分反应数据和外源或内源的同位素标肽(如果有),(iv) 得分考虑MS2谱图的高分辨及高精度质量信息和 (v) 得分来自于挖掘母离子(MS1)中蕴藏的信息。以上所有或大多数互相独立的打分或多或少的都会被以下软件所整合:OpenSWATH,Skyline,PeakView 和Spectronaut,并且会将它们组成成一个单一的最终判别打分。
分组打分和q-value评估
通过采用对SWATH-MS数据的提取离子色谱信息进行上节所说的独立打分,便能计算这些得分的分布。理想情况下,这些得分的“真阳性”和“假阳性”分布会清晰地分隔开。然后,有些得分可能因为实验相关的一些因素如样品复杂度,仪器表现和SWATH-MS参数设置等原因比其他一些得分更高,但单一得分并不足以判断肽段定性和定量结果的准确性。因此,将多种独立打分结果混合成一个单一的判别分数可以对肽段定性实现尽可能高的灵敏度(图5D)。我们可以用半监督学习算法来实现这一目的(Kall et al,2007),其采用循环逼近的策略得到独立得分间最优化的权重分配组合实现充分的分离target和decoy结果。我们通常认为判别得分最高的谱峰组(第一名,图5C)是最可能检测到的匹配肽段,并最终通过跨样品的结果比对来确认其准确性。SWATH-MS数据的成功学习和统计建模依赖于几个因素。首先半监督算法获得真实检测结果的得分特征并实现合理独立打分权重组合依赖于获取足够数量的高得分真阳性肽段。其次,decoy肽段序列必须合理的代表那些无法在此样品中检测到的肽段。综上,我们强烈建议分析完SWATH-MS工具中相关报告的target和decoy得分分布,并确认(i) 基于decoy得分分布得到的假设并无错误(比如正态性),(ii) target得分分布在分析复杂样品且谱图库相对规模较大时应该接近于双峰分布,(iii)decoy 分布的顶端,形状和宽度与target双峰分布中的真阴性部分匹配(图5D)。在某些情况下,谱图库如果和样品中检测到的母离子高度一致(> 90%),target分布的真阴性部分可能会非常小,从而导致要点(iii)难以判断(Reiter et al,2011)。如果有一个或更多要点无法满足,那么机器学习或q-value评估这一步可能就会失败或者产生偏差,从而导致统计结果失真及不合理的错误比例控制。
特定背景的错误率评估
上文描述的分析策略通常应用于单个样品检测。然后在某些情况下这样做会引入偏差,尤其是样品肽段数量巨大的同时分析对象数据集里包含了大量的异质样品或背景数据库里有大量的不同类型来源的样品信息(比如来自于公共数据库) 以及库里有可能包含大量对于检测样品来说的真阴性肽段和蛋白质。因此,我们建议根据特定数据的需求来设计分析策略,具体可以参考Rosenberger et al (2017a)。简略地说,为了保证整个数据集各个样品间的打分和统计结果的可比性,半监督的机器学习步骤需要在所有样品、所有候选谱峰组结果以整个实验为单位进行计算(图5E)。就优化实际体验来说(文件大小和内存限制),这步也可以考虑采用每个样品随机采用小部分肽段匹配结果进行统计评估来代表整个数据集的结果(Rosenberger et al,2017a)。1% 肽段FDR的阈值用于过滤每个单独的样品(参考图5E “run-specific” context)可能对小样本数据及样品专属的谱图库是足够有效地,但是在处理大规模样本的海量匹配结果时,假阳性可能就会累积。和DDA数据库搜库策略相似,大规模数据集上采用更严格的如多重假设检验的打分过滤策略是必要的。我们因此建议不仅仅在肽段水平,蛋白水平也要进行target decoy的假阳性评估(Rosenberger et al,2017a)。还有最好是采用基于“整个实验样品水平上”FDR评估,采用单一得分分布的q-value值,并且基于“global”策略,对所有样品的同一个谱峰只选取最佳匹配结果的肽段及蛋白组装信息。我们建议为了防止大样本量实验的假阳性率累积,最好采用全局蛋白及肽段的1%FDR控制(图5E; Rosenberger et al,2017a)。以上描述的基于特定背景的错误率控制方法目前在OpenSWATH的组件PyProphet(Rosenberger et al,2017a),和Spectronaut (Bruderer et al,2017)均能使用。
样品间对齐和定性置信度迁移
基于肽段中心打分算法的一个主要假设就是每个肽段只有最高得分的谱峰组能够反映其真实信号。实际上并不总是如此理想,比如当多肽丰度在一个或多个样本队列中下降到检测限以下时,非来源于目标肽的候选峰组得分较高。这个错的谱峰组检测和真正的肽保留时间不同,导致在最终的肽量化矩阵中出现一个错误值是经常发生的。通过再分析跨样本和保留时间的一致性以及用最可靠鉴定结果的样品来对齐谱峰组的峰边界,就可以检测并修改假谱峰组产生的排名错误(图5F)。该处理方式有时候可以重排谱峰组结果或挽回一些被1%肽段FDR过滤去除但其实落在预期保留时间内的结果。现有的多样品保留时间对齐的算法比如TRIC (TRansfer of Identification Confidence),目前是OpenSWATH的分析流程中的一部分(Rost et al,2016a)。
综上,多样品对齐方法和目前已经成功应用在DDA数据中国的相应分析方法类似(Prakash et al,2006; Mueller et al,2007; Cox et al,2014)。不过,DDA肽段定性和定量结果是基于保留时间和母离子m/z进行对齐而SWATH-MS会采用所有碎片离子作为对齐参考信息并控制错误率。
肽段和蛋白质定量
和其他自下而上的LC-MS/MS蛋白质组工具类似,SWATH-MS的定量信息是来自于肽段,通过多个碎片离子的峰面积进行加和或者平均得出。当然不是非得用肽段所有的碎片离子计算才能精确定量。最好选择高质量和信号没被干扰的碎片进行定量,当然目前全自动的分析算法已经开发成功(Keller et al,2015; Teleman et al,2015)。另外,基于母离子提取离子流的MS1定量方式可以提供正交的定量信息(Rardin et al,2015)。然后,在很多情况下,母离子定量方法在临近定量灵敏度下限(LLOQ)时,会比基于碎片离子定量的方法更不准确(Gillet et al,2012; Egertson et al,2013; Collins et al,2017)。这是因为MS1全扫描相对于MS2隔离窗口检测的选择性更低。为了从SWATH-MS中推导蛋白质的丰度,一个或多个肽段检测到的信号强度需要合计为最终每个样品中的蛋白质信号强度。为了实现该目标而开发了多种分析策略,比如:(i)每个蛋白质取其中最强信号的n个肽段进行加和或平均(Silva et al,2006; Ludwig et al,2012),(ii) 将所有肽段信号强度进行加和而不管其是否在每个样品都能检测到 (iBAQ; Schwanhausser et al,2011),或(iii)只考虑在比较的各个组别中均能检测到的肽段用于蛋白质的定量(LFQ; Cox et al,2014)。这些加和的蛋白质强度值可以根据需要进行归一化来反应各个蛋白的可检测性,比如根据蛋白长度(Zybailov et al,2006),理论肽段数(Schwanhausser et al,2011) 或计算得到的肽段检出可能性(Lu et al,2007)。缺失值可以通过多样品间的对齐分析(Rost et al,2016a) 或者统计算法插值来填补(Karpievitch et al,2012)。和通常仅仅检测一小部分目标蛋白的代表肽段来定量的SRM或PRM技术相比,以及大规模样品中每个蛋白能定量到的肽段不一定相同的DDA技术相比,SWATH-MS基于肽段中心的分析提供了一个更加一致且完整肽段定量信息用以蛋白质定量。在不同实验分组间通过计算碎片离子强度差异从而计算差异蛋白或肽段的统计工具目前也已有发展(Chang et al,2012)。为了让蛋白定量比较的一致性达到最高,我们建议系统性的采用SWATH-MS数据中每个蛋白质最稳健的碎片和肽段统一比较。为了选出这些碎片和肽段,我们需要获取每个肽段的碎片离子相对信号强度和每个蛋白的肽段相对强度信息并用于整个数据集,比如MSstats (Choi et al,2014) 或mapDIA (Teo et al,2015) 这两个用于统计倍数差异的工具。
翻译后修饰
基于肽段中心打分的SWATH-MS基于当然也可以和翻译后修饰(PTM)肽段的定量检测兼容。然而,SWATH-MS数据分析修饰肽段也有其难点,主要是MS2谱图中可能包含高度相似的相关肽段影响检测,比如非修饰肽段,骨架相同、修饰相同而修饰位点不同的肽段或者骨架相同而携带相同或相似的同位素修饰。这些肽段可能在同一个SWATH-MS隔离窗口并产生多个高得分的谱峰组。
和DDA分析一样,也有人开发了专门分析DIA和SWATH-MS数据中修饰肽的策略。其中最直接的方法就是建立修饰肽的PQP,比如从富集了修饰肽段建立的DDA谱图库中提取相应信息或者采用开放式修饰搜索策略(Na & Paek,2015)。有另一种分析策略依赖于用非修饰肽段的PQP去匹配修饰肽段并重点关注除了最高得分外的其余高得分谱峰组。那些额外的谱峰组有可能是和非修饰肽位于同一个或不同的隔离窗口内。此类策略目前被整合入MSPLIT-DIA (Wang et al,2015)和SWATHProphetPTM (Keller et al,2016) 并被发现当谱图与能决定修饰位点的碎片足够相似时就能成功分析(Toprak et al,2014)。最近另一个叫做“Inference of Peptido-Forms” (IPF) (Rosenberger et al,2017b)的算法将标准的Open-SWATH分析流程拓展到了修饰肽。IPF采用两步分析流程,第一步和标准的OpenSWATH流程很像,从DDA或DIA采集的谱图库中提取最高质量谱峰组的6个碎片离子并对其进行打分。这些transition需要和目标肽段序列匹配及修饰类型、数量所匹配,不过它们不需要精确匹配位点。在第二步中,IPF提取MS1中的母离子和理论预测的可以区分修饰位点的碎片离子XIC。这些信息用贝叶斯分层模型整合入一个单一的肽段构型置信度得分,并给每个检测到的谱峰组打分。而位点置信度据此进行重新评估,即使其并不存在于谱图库中。
综上,PTM-SWATH分析可以用于分析全细胞裂解液也可以用于特定类型修饰的富集样品,比如磷酸化富集样品。近年来,已有多个修饰类型被成功进行SWATH-MS分析 (Krautkramer et al,2015; Sidoli et al,2015; Lawrence et al,2016; Rosenberger et al,2017b)。尤其是包含大量不同状态或样品重复的实验,我们也可以将类似的定性迁移和定量分析策略用于分析修饰和非修饰肽段。
展望
自从21世纪初最早期的DIA应用至今(Masselon et al,2000; Purvine et al,2003; Venable et al,2004),仪器端的数据采集速度,质量精度和分辨率获得了巨大的改进。同样的,从肽段中心打分算法提出至今(Gillet et al,2012),谱图库的建立(Schubert et al,2015a),自动分析流程(Rost et al,2014,2016a; Keller et al,2015)和肽段及蛋白质错误率统计控制 (Rosenberger et al,2017a)等分析方法也得到了极大的改进。SWATH-MS数据采集的高可重复性及精确度已被全世界方位内各个实验室的一系列研究所证明(Collins et al,2017)。而且近期我们也对五个主流的基于肽段中心的数据分析工具进行了比较评测(Navarro et al,2016)。SWATH-MS的发展同样奠定了新型大规模生物研究的基础,比如,研究线粒体与小鼠肝脏代谢的关系 (Williams et al,2016),癌细胞药物治疗相应的磷酸化蛋白质组和染色质特征的研究(Litichevskiy et al,2018)或不同性状的遗传和环境对同卵和异卵双胞胎贡献研究(Liu et al,2015)。在本教程中,我们记录了SWATH-MS实验在建立工作流、软件工具和分析流程中的一系列进展,希望SWATH-MS在未来几年支持和加速更多的研究。尽管迄今为止大多数出版的SWATH-MS研究都使用DDA数据构建的谱图库,但已有新的方法不依赖库了。谱图为中心的打分策略诸如DIA-Umpire (Tsou et al,2015)或Group-DIA (Li et al,2015)从DIA数据直接构建虚拟MS2谱图并用常规的数据库检索方式分析。肽段中心且不用谱图库的分析工具如FT-ARM (Weisbrod et al,2012) 或PECAN (Ting et al,2017)则利用理论碎片预测并匹配SWATH-MS数据中的多通道MS2谱图。有趣的是,依靠先验知识的谱图库的分析工具相比library-free工具能更好地应对选择性低的数据(Navarro et al,2016; Ting et al,2017)。这一发现表明,随着新仪器能够提供选择性的提高更好的数据(更小的母离子隔离窗口,更高的色谱峰容量,更好的四级杆或ion mobility分离效果等),先验知识对数据分析可能会变得不那么重要了。但另一方面,以不同器官的深度蛋白质组覆盖数据构成的先验知识库或者合成蛋白质组学在同时也有可能得到大幅提升。这可能会刺激开发新的分析方法,从而更好地利用这些库。
我们希望未来的质谱仪在灵敏度和扫描速度将继续改善。可预见的是, 采集高质量的母离子和碎片离子谱图所需的必要将减少,从而允许更多较窄的隔离窗口,使隔离窗口宽度会接近常用的DDA和PRM中使用的1 - 3 m/z。我们推测,仪器灵敏度和速度的提高将达到一个阈值,最终通过单一的“超级”方法就可以实现目前使用的所有数据采集方法的优点。接下来的问题将不再是一个是否应该做一个DDA,DIA或靶向实验,而是是否使用肽段中心peptide-centric还是谱图中心spectrum-centric数据分析策略。但采用谱图中心spectrum-centric方法(其中每个连续扫描一份独立进行数据库搜索的算法)似乎不太可能达到最好的效果,因为色谱维度中的丰度的信息和之前的碎片信息不会被利用到。因此,更有可能是要么肽段中心或混合方法谱图中心和肽段中心的方法能够最好的利用未来高度复杂和选择性的数据集。未来其他DIA,尤其是SWATH-MS所需要具备的特性则是更简便的数据采集方法和高通量蛋白质组学的适用性。目前,大约50%的质谱可检测的蛋白质组可以在相对较快的时间尺度(每样本< 1小时)通过SWATH-MS重复测量。这一进步将使得未来蛋白质组学数据的获得变得更加大众化,更能够方便的进行多类型交叉研究,而这正是我们一直以来希望克服的困难。
参考文献
1. Abbatiello SE, Schilling B, Mani DR, Zimmerman LJ, Hall SC, MacLean B, Albertolle M, Allen S, Burgess M, Cusack MP, Gosh M, Hedrick V, Held JM, Inerowicz HD, Jackson A, Keshishian H, Kinsinger CR, Lyssand J, Makowski L, Mesri M et al. (2015) Large-Scale Interlaboratory Study to Develop, Analytically Validate and Apply Highly Multiplexed, Quantitative Peptide Assays to Measure Cancer-Relevant Proteins in Plasma. Molecular & cellular proteomics : MCP 14: 2357-2374
2. Addona TA, Abbatiello SE, Schilling B, Skates SJ, Mani DR, Bunk DM, Spiegelman CH, Zimmerman LJ, Ham AJ, Keshishian H, Hall SC, Allen S, Blackman RK, Borchers CH, Buck C, Cardasis HL, Cusack MP, Dodder NG, Gibson BW, Held JM et al. (2009) Multi-site
