Technical Data
技术资料
搜库用的计算机的成本预算相对于质谱来说忽略不计,但在相对固定预算下配备最合适的计算机又何乐而不为呢?
为何现在做这个内容?
2017年注定对于我们做质谱相关软硬件解决方案的公司来说需要好好学习行业动态:AMD在苏妈带领下终于发布了高性价比的桌面级别的CPU,Intel被逼无奈狠狠的在便秘了将近5年后挤了一大管牙膏发布了重大更新8代桌面CPU及全新i9处理器(新一代志强GOLD系列对于蛋白质组分析来说性价比不高,不在本文中讨论)。同时内存和SSD在短短两年间价格翻番,再用1、2年前的老配置给客户做方案显得如此不合时宜。
对于近期有采购质谱配套计算机、升级旧平台计算机、采购全新分析软件的研究者,可以参考本文对原有系统进行测试进行对比。
相关下载
链接:http://pan.baidu.com/s/1nv0xweP 密码:iilc
其中包括:
1. 测试用RAW文件及原始文献(如需该文献所有RAW可自行去PRIDE下载或联系我们)
2. Fasta数据库:Uniprot人,Ncbi 脊椎动物
3. Maxquant及参考用参数文件
4. 评估计算性能用软件:CPU-Z, Cienbench R15
计算机硬件有哪些和搜库相关?
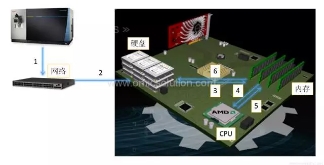
1. 网络传输,建议千兆局域网起步,有条件的可以采用万兆方案,在实验室外网上传速度不超过200Mbps前不建议采用云计算方案
2. 数据保存,目前常规硬盘规格为8-12TB,基本满足大数据量存储需求,但高性能SSD近期价格处于高位,建议系统及分析软件使用500-1TB NVMe SSD
3. 内存:重要计算缓存部件,建议16GB起步,大数据量、大数据库分析进一步提升至64GB以上,内存速度建议2666MHz以上即可。
4. CPU:最重要的计算部件,后文具体讨论
5. 显卡:非重要部件,支持1080P以上分辨率显示即可
6. 其他部件均不对计算过程产生重要影响
质谱数据分析各步骤相关的硬件

与搜库速度相关的软件设置
1. 软件差异:(Maxquant、PD、Mascot、PEAKS、pFind)本身算法不同,参数不同,功能不同,没法绝对比较;
2. 数据差异:RAW,MGF,保存格式是Profile还是centroid;
3. 软件分析过程是否支持多核心;
4. 参数:容差、酶切特异性、修饰、定量方法;
以上参数不在本次讨论中,故我们选择相同软件及搜库参数进行比较,忽略软件及参数差异,主要比较CPU 差异及数据库、RAW文件数量对搜库的速度影响
示例数据
软件
Maxquant 1.6.0.16
OS: Windows 10 x64
数据
MCP Multiplexed, Quantitative Workflow for Sensitive Biomarker Discovery in Plasma Yields Novel Candidates for Early Myocardial Injury Supermix 去高丰度,ITRAQ 4PLEX 数据集Patient 1,含30个QE的raw 分级数据,每个分级检测了180分钟,测试分两部分:第一部分为全部30个分级搜库,第二部分为选择前3个分级搜库。 Maxquant参数: 数据库为Uniprot Human Proteome 201611数据库 70957个蛋白,MS2 定量,开启Filter by PIF,其余全部默认,开启所有可用计算核心
硬件

相关说明
CPU多核心:单个CPU包含n个处理核心,每个核心为独立的物理单元,可以独立执行一个分析任务
超线程(HT):将单个核心虚拟成多个线程,使得计算机在处理大量任务时的效率得到提升。但其只是一种模拟多核心的技术,并没有实际提升CPU计算能力。在任务数量较少,而单个任务只支持单线程时由于无法利用整个核心的计算能力,速度反而大幅下降。蛋白质组学数据特性使得HT技术用处不大,本次测试默认均关闭。
内存:CPU进行计算时会依次读取其缓存的数据进行计算,但缓存容量非常小,绝大多数数据都会临时存放在内存中等待计算,因此内存容量和速度会影响CPU的计算效率,通常建议CPU核心数量*4=内存G大小。内存的速度简单地说目前主流在2133~3000,3000MHz的内存相对于2133MHz的内存对计算性能有大约10%的提升。由于我们手头没有多种型号内存,故本次测试不对内存速度进行比较。
搜库中各个步骤分别占用的时间

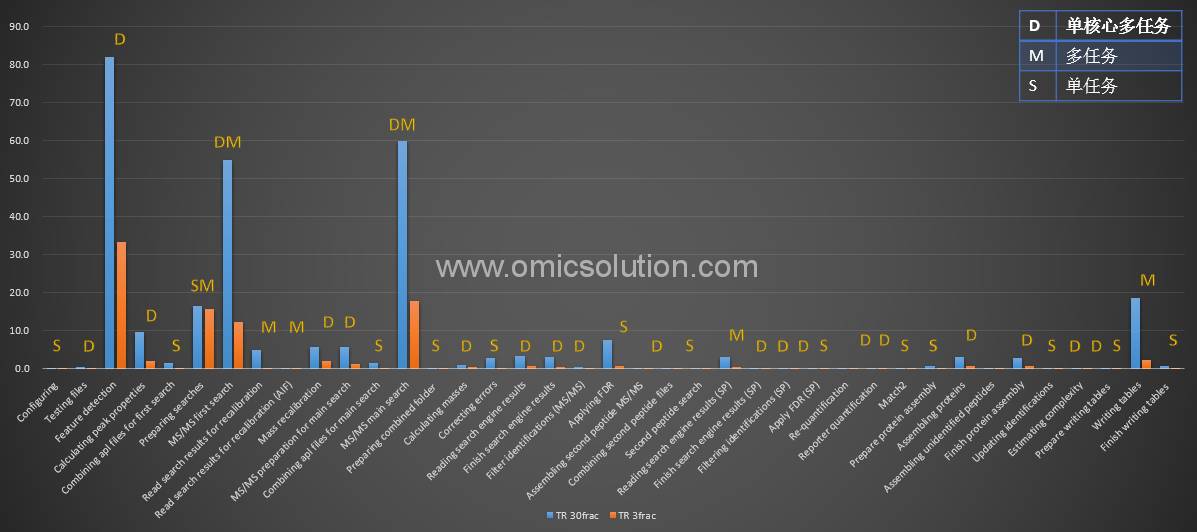
结论:
1. 同时处理的任务越少,速度越取决于CPU主频;
2. 同时处理任务越多,速度越取决于核心数量,但不完全成正比;
3. 相同主频的CPU,制程越先进的越快;
4. 内存满足基本要求即可,高低频率速度差别在10%左右(数据未列出);
5. 硬盘用SSD肯定可以提升性能,但不同规格SSD对搜库影响不大
数据库大小对搜库速度的影响间
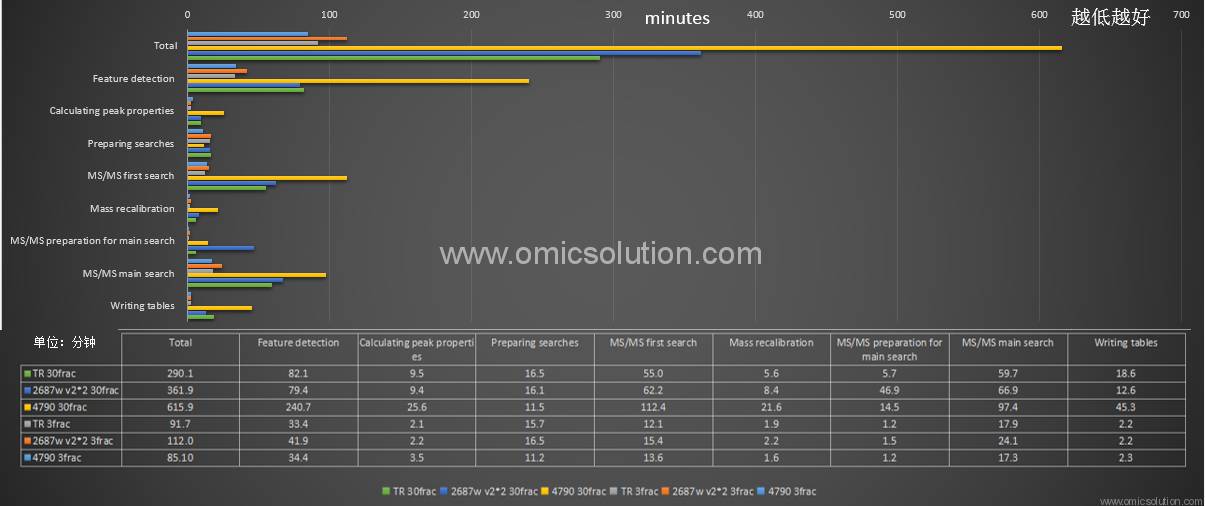
保持其他参数不变的情况下,我们采用TR平台 3个frac ITRAQ human数据,将数据库更换成NR metazoa全库(约24000000个蛋白)和Uniprot proteome human(约70000个蛋白)进行比较。以下列出有显著差异的计算步骤。
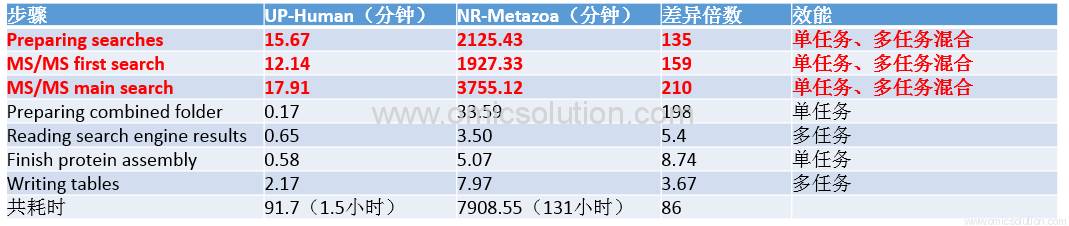
通过对整个搜库过程的观察,在最为耗时的三个步骤中Maxquant均无法保持全部分析流程的多任务处理,比如在搜索过程中,虽然搜索可以充分利用所有线程进行,但是在搜索完毕一部分数据后需要进行整合再分发,这个过程却是单任务模式的,往往耗时长久成为瓶颈。数据库准备过程也是一样,多任务的步骤快速完成后就需要等待单任务的步骤进行长时间的计算,且同时观察到最高内存占用达到了40GB以上。
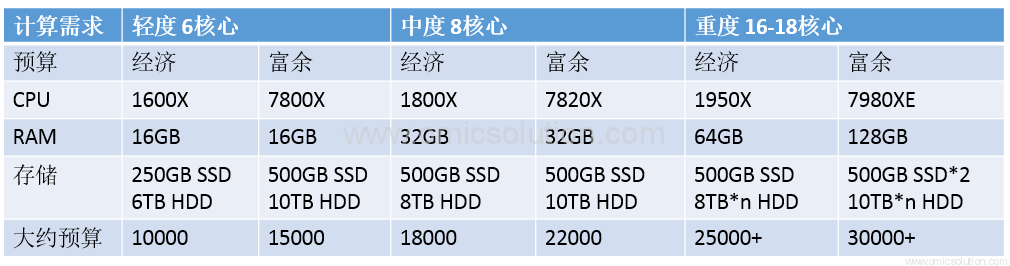
附表:硬件介绍

附表:性价比
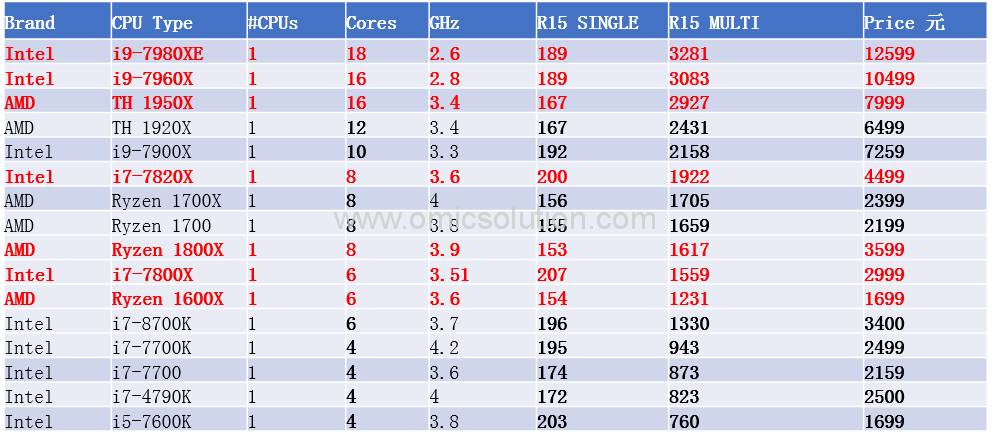
除非任务非常繁重,同时需要分析的RAW文件非常多,同时预算非常多,否则不推荐上志强多路CPU,性价比不高
结语
1. 在2016年的计算机硬件没有特别适合质谱分析的桌面硬件配置,故我们都会采用志强系列工作站或者服务器,但现在,新一代的硬件上市后我们可以和又贵又难维护升级的高级货们说再见了。
2. 以下是推荐的配置,可以根据实际价格和需求调整,更高配置的AMD EPYC和INTEL XEON GOLD在搜库时性价比并不好,仅推荐大量多任务需求的研究者使用。单一物种搜库为主要任务时对内存无高要求,经常进行大数据库搜索时建议增加内存数量配置。
3. 有大数据库搜索需求的研究者必须考量CPU单核性能同时配备大内存,否则会大大拖累计算时间(请注意关闭HT)
4. 如果您既有大量数据又需要分析超大规模数据库的分析需求,那么7980XE系统可能是目前较好的解决方案,不建议选择志强等工作站、服务器平台,这些平台的单核性能孱弱,可能完全发挥不出其应有的性能。