Technical Data
技术资料
“de novo”一词源自于拉丁文,在英文中的意思是“over again”或者是“anew”。对于de novo peptide sequencing的通俗定义则为在不知道理论氨基酸序列的情况下,对多肽/蛋白进行从头测序。我们熟知的经典的Edman降解法通过依次降解经人为修饰后的N末端氨基酸,从而实现对于多肽/蛋白N-端长度达60-70个AAs (amino acids) 的测序。这样的工作是十分耗时耗力的,虽经自动化后相比之前节省了大量的人力和时间成本,但由于其本身原理所限(1. 反应效率达不到100%;2. 产物不可能无限回收循环),这种方法势必满足不了日渐旺盛的蛋白测序需求。
在高灵敏度、高分辨率质谱仪器的不断更新换代和算法的日趋优化下,基于MS/MS数据的de novo peptide sequencing也在逐步发展和完善中。其基本原理是通过计算相邻碎片离子之间的质量差来解析出肽链上的每一个氨基酸残基。例如,在常见的CID串级谱图中,能够得到大量b、y碎片离子信息,相邻的y离子或b离子之间的质量差则为其中某一个氨基酸残基的分子量(如图1示例,y11与y10之间相差101.0415 Da,为苏氨酸残基(Threonine, T)的分子量,可点击此链接查询单个或者组合氨基酸残基分子量表格)。在极其理想的情况下,如果某条肽段的串级质谱图上鉴定到了所有连续的y离子或者b离子,那么我们就能解析出这条肽段的氨基酸序列信息。
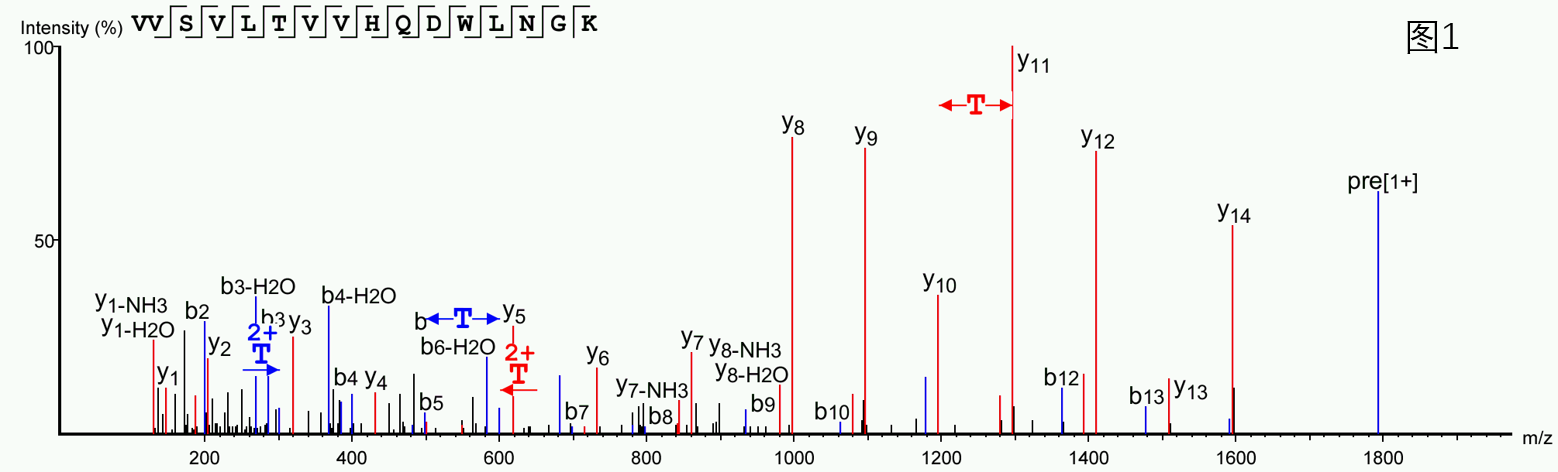
然而,理想很丰满,现实很骨感。很多时候,由于客观条件的种种限制,我们拿到的二级谱图不可能那么完美(某些碎片离子的信息可能丢失),除了b、y离子之外还有其它一些类型离子的存在(如图1中的y1-NH3,y8-H2O等),肽段本身还存在各种各样的翻译后修饰,这些因素都会进一步加大谱图的解析难度。如果纯靠人力来进行这一工作,估计十个人中会有九个会崩溃,剩下一个在拼命解谱。好在这个世界只要有需求,总会有人想出办法来简化工作流程,提高工作效率,促进人类进步。早在2002年,Bioinformatics Solutions Inc. (BSI) 发布的第一款PEAKS Studio software即以全自动化的de novo peptide sequencing为基础,对于传统的搜库流程进行了改革和创新,使高通量蛋白质组学实验产出的海量MS/MS数据被尽可能有效的利用,极大程度地提高了谱图鉴定率和搜库结果的准确性。
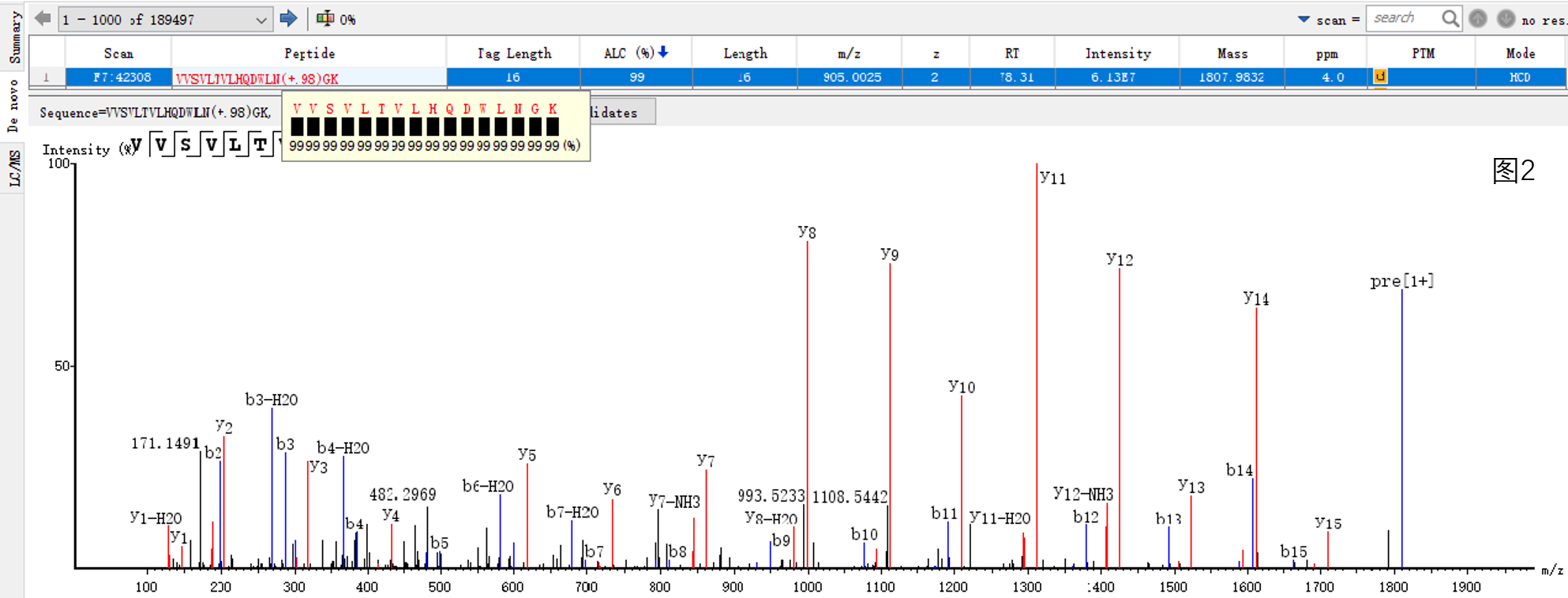
在最终的PEAKS Studio报告结果中,对于那些在数据库中无法得到正确匹配、也排除了各种翻译后修饰可能性的高质量谱图,会有包含每个氨基酸残基位点置信度得分在内的十分详细的肽段de novo结果(如图2示例)展示,这就带给质谱研究工作者极大的便利:以往我们只能从旧有的数据库中发掘我们的样品信息,现在我们不仅能够知道样品中未知肽段的存在,并且,在de novo结果的辅助下,还可以进行反推和地毯式的搜寻,对所研究的对象有了更加深入的了解。Mass Spectrometer,不再是外行人眼中高精度的“电子天平”,而是分析界极其powerful的discovery型仪器。如果说分析人员从样品处理、蛋白酶解到色谱分离、质谱检测都已经做到了尽善尽美,那么,最后的数据挖掘过程,则是决定我们的实验结果是否出众,抑或是平庸的关键因素。
不得不再提一下,PEAKS Studio在de novo peptide sequencing方面有非常多成功的案例。如果各位看官有类似的需求,不妨写邮件到support@omicsolution.com,咨询我们的技术客服,对于您的具体研究对象,我们会提供一对一的订制化实验方案,为您解决肽段从头测序的难题!
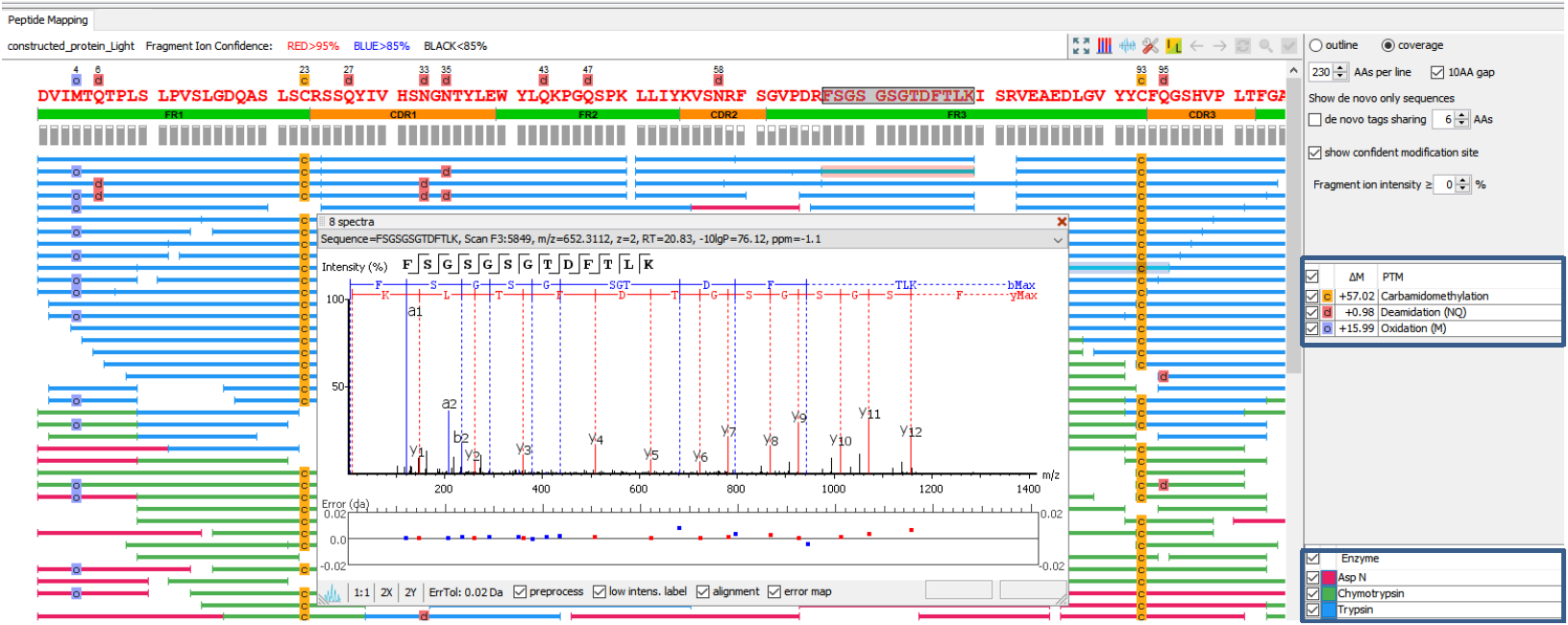
话说回来,让我们回到今天的主题——抗体蛋白的从头测序。前面我们提到了,PEAKS Studio能够在肽段层面上给出十分可信的de novo结果。可是,究竟肽段和肽段之间是怎么相连的,还需要质谱专家们凭借自己丰富的解谱经验来进行后期的人工拼接,这个工作量同样非常巨大。幸好,Bioinformatics Solutions Inc. (BSI) 发布的这一款PEAKS AB软件给生物制药界的同仁们带来了福音:在不提供理论氨基酸序列的情况下,PEAKS AB可以百分百实现抗体蛋白全长序列的de novo解析。惊喜不惊喜,开心不开心!可以这么说,PEAKS AB是借助PEAKS Studio强大的de novo peptide sequencing解析能力,为抗体蛋白量身定制的一款专业软件。前一期的主题我们也提到了,传统的单克隆抗体药物表征解决方案必须要知晓所要分析的抗体药物的理论氨基酸序列且保证其准确性,然而,PEAKS AB除了可以提供传统思路的已知蛋白序列确证的搜库方案,还能在毋需提供参考序列的情况下实现未知蛋白的准确测序。用户在将原始数据导入PEAKS AB软件后,只需键入寥寥几个参数,即可得到如图所示的从肽段水平拼接的抗体蛋白一级结构信息,包含序列信息、CDR区标识、精确到氨基酸残基的位点置信度、翻译后修饰信息。点击每一条supporting peptides,还可以看到其二级谱图及碎片离子匹配、质量偏移等。是不是你想能想到的,应有尽有!